微生物インタラクション(渡辺研究室)
-
研究室ホームページ
- https://bsw3.naist.jp/microbial_interaction/
-
Webメディア
NAIST Edge BIOは、奈良先端科学技術大学院大学 バイオサイエンス領域 の各研究室で取り組んでいる「最先端」の研究プロジェクトや研究成果について、研究者だけではなく受験生や一般の方にも分かりやすく紹介するためのWebメディアです。 - NAIST Edge BIO 第23回
研究・教育の概要
人類に身近な微生物がどのようにふるまい、他生物や環境要因とどのように相互作用することで複雑な生態系を構築するのかを分子・代謝・細胞レベルで明らかにし、ミクロの世界における多様性の理解を目指した研究・教育を行います。食と健康を意識したバイオ技術にも貢献していきます(図1)。
主な研究テーマ
伝統的発酵食品における微生物生態(図2)
発酵食品のおいしさや健康機能性は国内外で大きな注目を集めており、その需要は高まっています。私たちは、発酵食品とそこに暮らす微生物たち(酵母、乳酸菌、麹菌など)への理解を深めることで、次世代に向けた食の未来を切り開いていきたいと考えています。人間の目には見えないミクロな世界での微生物のふるまいや相互作用には、いまだに多くの謎が残されています。特に、日本酒、醤油、漬物のような昔ながらの伝統的発酵食品に見られる、複雑な微生物生態系がどのように構築され、機能しているのかを探究しています。また、かつて古の“発酵の都”でもあった奈良の地に残る酵食品に注目した独創性の高い研究にも挑んでいます。これらの研究を通じて、伝統的な知見を活かしながら、微生物生態系の構築原理や環境適応メカニズムを解明し、発酵という現象の本質に迫ることを目指しています。
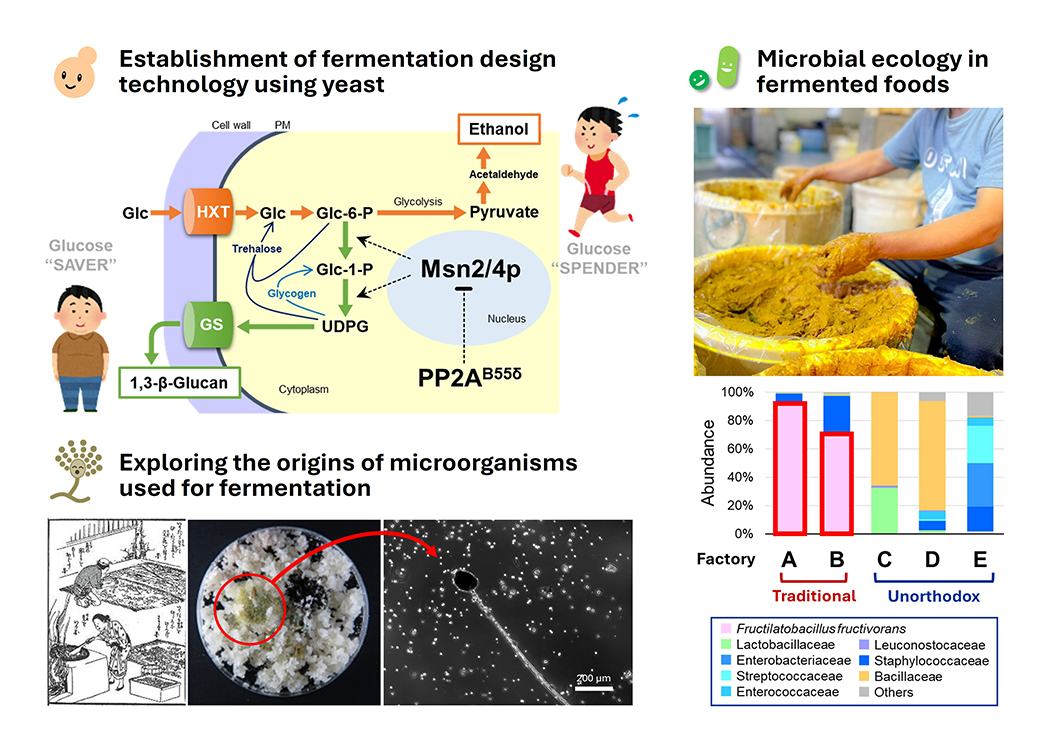
(図2) 私たちは、日本酒の醸造で用いられる酵母の研究をきっかけに、アルコール発酵デザイン技術の確立を目指しています(左上)。また、発酵食品に関与する微生物のルーツを調べるため、自然界などからの探索を進めています(左下)。奈良漬のような伝統的発酵食品に潜む未知の微生物生態系の解明にも挑んでいます(右)。
麹菌に由来する発酵食品の健康増進作用(図3)
日本では古来、多種多様な伝統的発酵食品が造られ、現代に至るまで私たち日本人の食生活および健康長寿を支え続けています。しかしながら、発酵食品の健康増進作用に関しては科学的に未解明な点が多く残されています。私たちは、発酵食品の健康増進作用を微生物学的見地から解明しようとしています。特に原料分解酵素の供給源として、様々な発酵食品製造の要を担う麹菌に着目し、麹菌が生産する機能性物質探索およびその生産機序解明を目指しています。近年、麹菌が「生活の質(QOL)」改善作用を有する機能性ポリアミンの一種・アグマチンを著量生産することを明らかにしました。麹菌によるポリアミン生産の分子機序解明に加え、得られた知見を基に発酵食品の高機能化や高齢社会における健康長寿の促進に資する技術開発にも取り組みます。
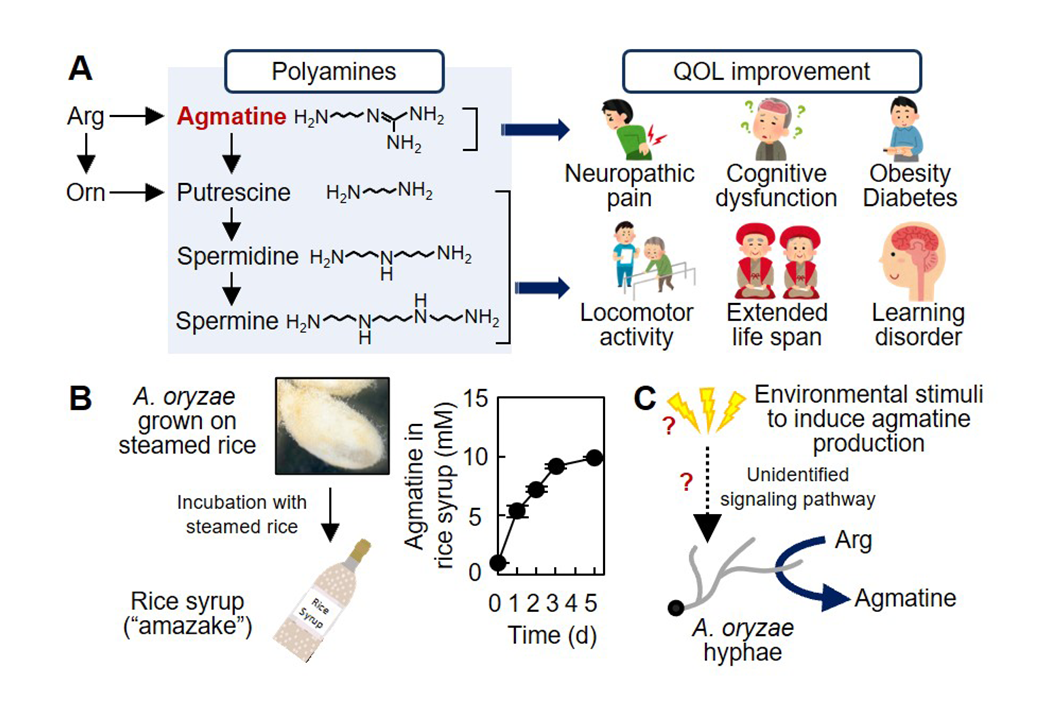
(図3) ポリアミン生合成経路(A、左)。アグマチンを含むポリアミンは様々なQOL改善効果を有することが知られています(A、右)。蒸米を基質として麹菌を培養すると、糖化液(麹甘酒)中に著量のアグマチンを生産・蓄積することを明らかにしました(B)。最近では、アグマチン生産の分子メカニズム解明を目指し研究を行っています(C)。
TOR (Target of Rapamycin)シグナル経路の解明(図4)
TORキナーゼは、TORC1、TORC2と呼ばれる複合体を形成し、栄養源や成長因子などの刺激を伝達するシグナル経路で働きます。これらの活性異常は、ガン化や糖尿病だけでなく、老化や寿命にも深く関わることも明らかになり、医薬分野におけるターゲットとして注目されています。私たちは、ヒトと類似のTOR経路をもつ分裂酵母をモデルとして、多面的なアプローチによりその全貌を分子レベルで明らかにしようとしています。また我々は、細胞増殖を促進するTORC1が高温では増殖を抑えることを明らかにしています。地球温暖化が懸念される昨今において、高温環境における増殖制御メカニズムの理解は非常に重要です。また、酵母の高温増殖能の理解はバイオエタノールの生産コスト削減にも有用であり、その普及への貢献も期待されます。
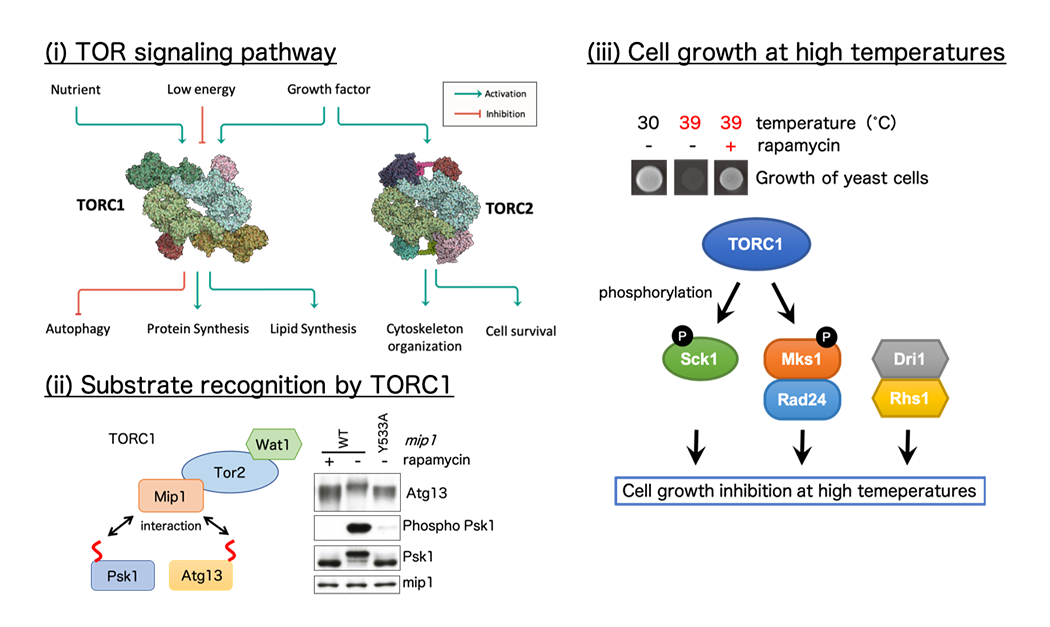
(図4) 栄養情報を伝達するTORシグナル経路 (i)。分裂酵母をモデルとしてTORC1の基質認識メカニズムを解明しました (ii)。また最近では、高温環境における細胞増殖の抑制機構に興味を持って研究を進めています (iii)。
主な発表論文・著作
- Morozumi et al., FEBS Lett., 598, 2886-2896, 2024
- Murakami et al. Appl. Environ. Microbiol., 90, e0029424, 2024
- Morozumi et al., iScience, 27, 108777, 2024
- Watanabe et al., Int. J. Mol. Sci., 25, 304, 2024
- Tai et al., Mol. Cell Biol., 43, 675-692. 2023
- Watanabe et al., NPJ Sci. Food, 7, 37, 2023
- Watanabe and Hashimoto, Sci. Rep., 13, 9279, 2023
- Morozumi et al., J. Cell. Sci., 134, jcs258865, 2021
- Ishii et al., J. Bacteriol., 203, e0016221, 2021
(こちらのリンクはすべて外部サイトへ遷移します![]() )
)
 奈良先端科学技術大学院大学
奈良先端科学技術大学院大学