バイオエンジニアリング(加藤晃研究室)
-
研究室ホームページ
- https://bsw3.naist.jp/ko-kato/
-
全学オンラインセミナー
2022年度に開催されたBio Discovery Session(全学オンラインセミナー)のアーカイブ動画の一覧です。 - Bio Discovery Session
-
Webメディア
NAIST Edge BIOは、奈良先端科学技術大学院大学 バイオサイエンス領域 の各研究室で取り組んでいる「最先端」の研究プロジェクトや研究成果について、研究者だけではなく受験生や一般の方にも分かりやすく紹介するためのWebメディアです。 - NAIST Edge BIO 第24回
研究・教育の概要
バイオテクノロジーによる社会貢献を念頭に、バイオ医薬品などの有用タンパク質を植物で高生産するための基盤技術の開発や、ストレス耐性や環境適応などの表現型を制御する仕組みの解明を行っています(図1)。
研究室に配属された学生自身が研究を論理的に理解し、知識の裾野を広げ発展させていけるように指導を行っています。定期的な研究室ミーティングに加えて、産業界から研究者・実務者を招き、企業での研究開発に必要な知識を紹介します。これらの指導を通して、低成長・グローバル社会で幅広く活躍できる人材の育成を目指しています。
主な研究テーマ
導入遺伝子の高発現化技術の開発
植物を宿主とした、医療用ワクチンや成長因子などの有用タンパク質の生産は、ウィルスやプリオンの混入リスクの低さなどから非常に注目を集めています。一方で、生産性の低さが問題になることも多く、導入した遺伝子からタンパク質が生産されるまでの複数の過程を効率化することが求められています(図2)。そこで当研究室では、(1) 次世代シーケンサーと最適化アルゴリズムを用いて、転写に関わるコアプロモーターの解析や、mRNAの安定性や翻訳効率に関わる5’UTR配列や3'UTR配列を解析し、既存の遺伝子発現効率を超える全く新しい配列を設計するシステムの開発、(2) 標的RNA配列に特異的に結合する合成タンパク質を作製し、これに転写や翻訳、スプライシング制御に寄与する制御タンパク質を連結した人工調節因子を設計・利用することで標的遺伝子RNAを質的/量的に制御するシステムの開発、の2つのアプローチを通して植物細胞を対象に安定かつ生産性の高い物質生産システムの実用化に取り組んでいます。これまでに得られた成果は、複数の企業に提供し、既に一部の成果は実用化されました。
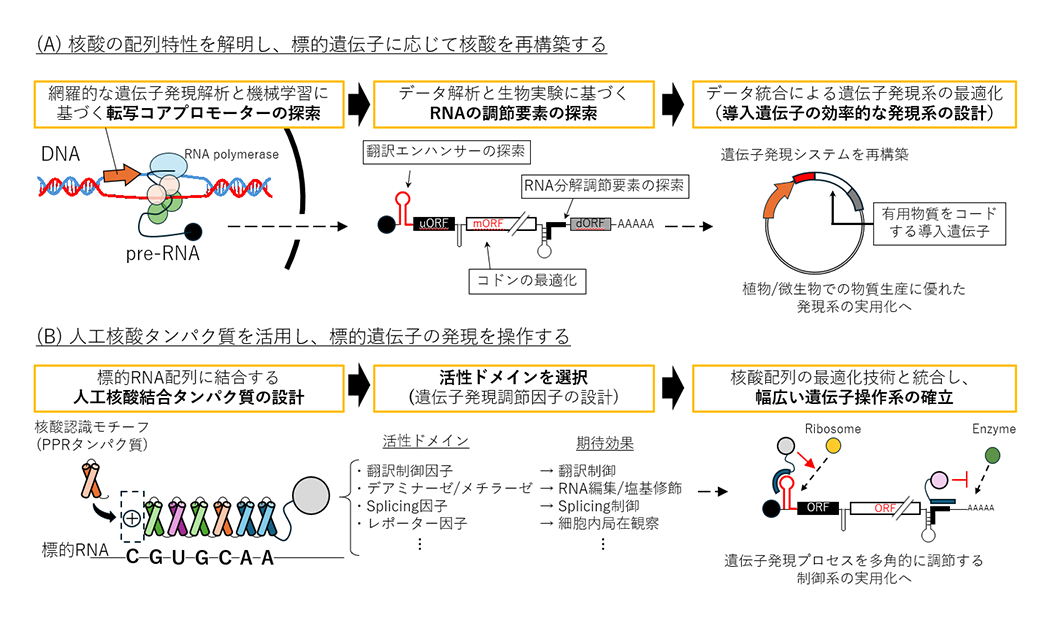
(図2) 導入遺伝子の高発現化技術の開発
(A) 網羅的な遺伝子解析と機械学習により、発現調節に寄与する配列要素を解明します。得られたデータを基に、導入遺伝子を効率的に発現させる最適化ベクターを構築し、有用物質生産のための発現系として利用する技術の開発を目指しています。
(B) 標的RNAの塩基配列に合わせてRNA結合タンパク質を人工合成し、その末端に遺伝子発現調節で働く因子の活性ドメインを連結することで、標的RNAの発現を人為的に制御する人工調節因子の開発と、実用化を目指しています。
遺伝子発現による表現型制御機構と適応進化機構の解明
遺伝子発現はその個体の表現型に直結します。この研究では、特に開花時期などの植物の繁殖成功にとって重要な形質に焦点を当て、ゲノム配列情報や遺伝子発現データに基づいて、表現型を制御する遺伝的要因をゲノム網羅的に検出することを試みています(図3)。また、検出された遺伝子がどのように表現型に関連しているかを明らかにします。野生植物の繁殖成功度を制御する仕組みや適応進化機構の理解、さらには将来的には農業への応用を目指しています。
遺伝子発現による植物の環境適応機構の解明
この研究では、植物が環境ストレス状態に巧みに耐える能力と、環境変動に柔軟に適応する仕組みに着目します(図4)。環境適応性の異なる系統を利用し、ゲノム解析により責任遺伝子を探索します。高解像度の遺伝子発現解析により、環境変動に対する応答機構の解明を目指します。
主な発表論文・著作
- Wakabayashi et al., Plant Biotechnol., in press
- Hattori et al., LSSR., in press
- Ueno D. et al., J. Biosci. Bioeng., 143, 450-461, 2022
- Ueno D. et al., Plant Sci., 318, 111241, 2022
- Han Q. et al., AoB Plants., 13, plab039, 2021
- Yagi N. et al., Nat. Commun., 12, 3684, 2021
- Ueno D. et al., BMC Bioinform., 22, 380, 2021
- Matsui T. et al., Plant Biotechnol., 38, 239-246, 2021
- Ueno D. et al., Plant Cell Physiol., 62, 143-155, 2021
- Shah N. et al., Nat. Commun., 11, 253, 2020
(こちらのリンクはすべて外部サイトへ遷移します![]() )
)
 奈良先端科学技術大学院大学
奈良先端科学技術大学院大学






