分子医学細胞生物学 (末次研究室)
-
研究室ホームページ
- https://bsw3.naist.jp/suetsugu/
-
全学オンラインセミナー
2022年度に開催されたBio Discovery Session(全学オンラインセミナー)のアーカイブ動画の一覧です。 - Bio Discovery Session
-
Webメディア
NAIST Edge BIOは、奈良先端科学技術大学院大学 バイオサイエンス領域 の各研究室で取り組んでいる「最先端」の研究プロジェクトや研究成果について、研究者だけではなく受験生や一般の方にも分かりやすく紹介するためのWebメディアです。 - NAIST Edge BIO 第7回
研究・教育の概要
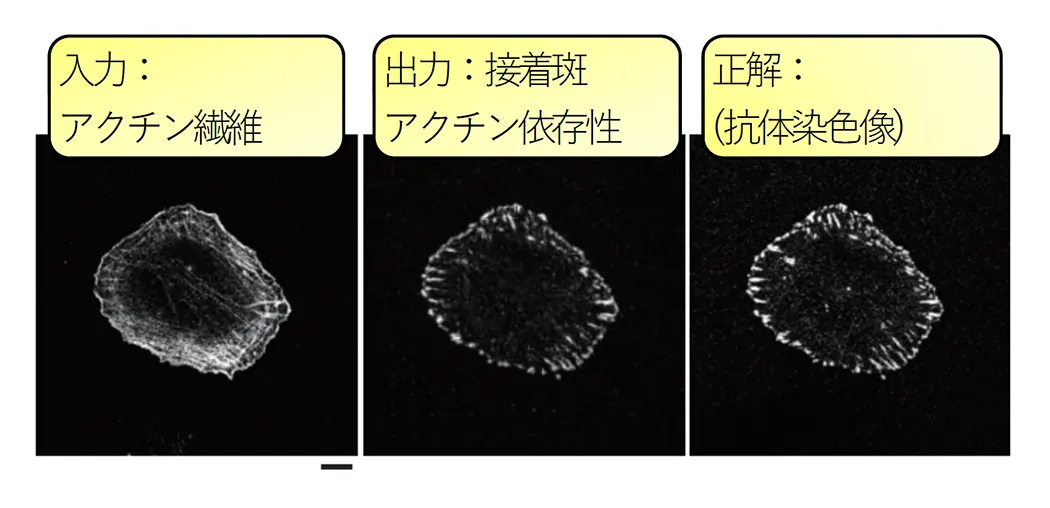
細胞膜は細胞の内外を区切り、細胞が生命として成り立つために必要不可欠な構造物です。細胞膜は、細胞の受ける様々な刺激を受容するために重要であると思われます。しかし、その細胞膜とその結合タンパク質のどのような組み合わせが、細胞を形作り、また、刺激に対する応答を可能にしているのか、不明な点が多く残されています。我々の研究室は、増殖と形態的な変化において細胞膜を細胞内シグナリングに接続している膜結合性タンパク質を研究します。細胞膜を構成する脂質分子の性質もまた、膜結合性タンパク質を使って調べられます。さらに細胞の形態を示す画像や、上記タンパク質の局在を示す画像を用いて、細胞の形態変化である細胞の振る舞いの解析を深層学習により行います。
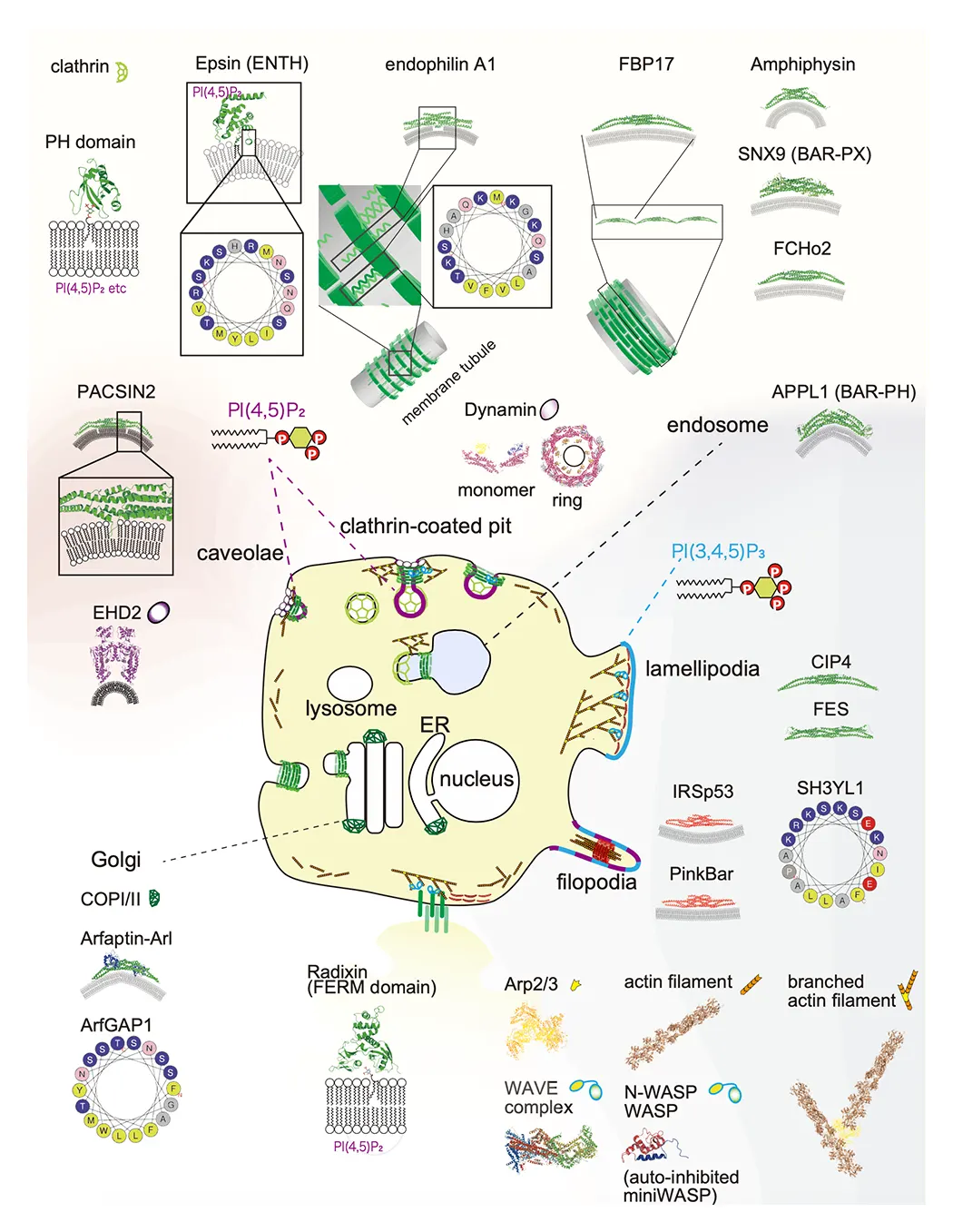
(図1) 研究室で対象としている細胞の形態形成を担うタンパク質(アクチンやBARドメイン、F-BARドメイン、I-BARドメインなどの膜結合タンパク質)と脂質の例(Suetsugu et al., Phys Rev 2014より)。
BARドメインは、突起(糸状仮足と膜状仮足を含む)及び、ミクロン以下のスケール陥入部(例えばクラスリンで被覆した穴とカベオラ)のポリマーとして機能し、微細構造を形成します。クラスリン被覆小孔とカベオラのための典型的大きさは、それぞれ、直径100-200nmと、直径50-100nmです。 BARドメインは、3-6nmの直径の20-25nmの弧として近似できます。 膜の厚さは、およそ5nmです。
主な研究テーマ
細胞膜の形態を形成するタンパク質と細胞膜の形態に依存した細胞内シグナル伝達、特に細胞のがんとの関連
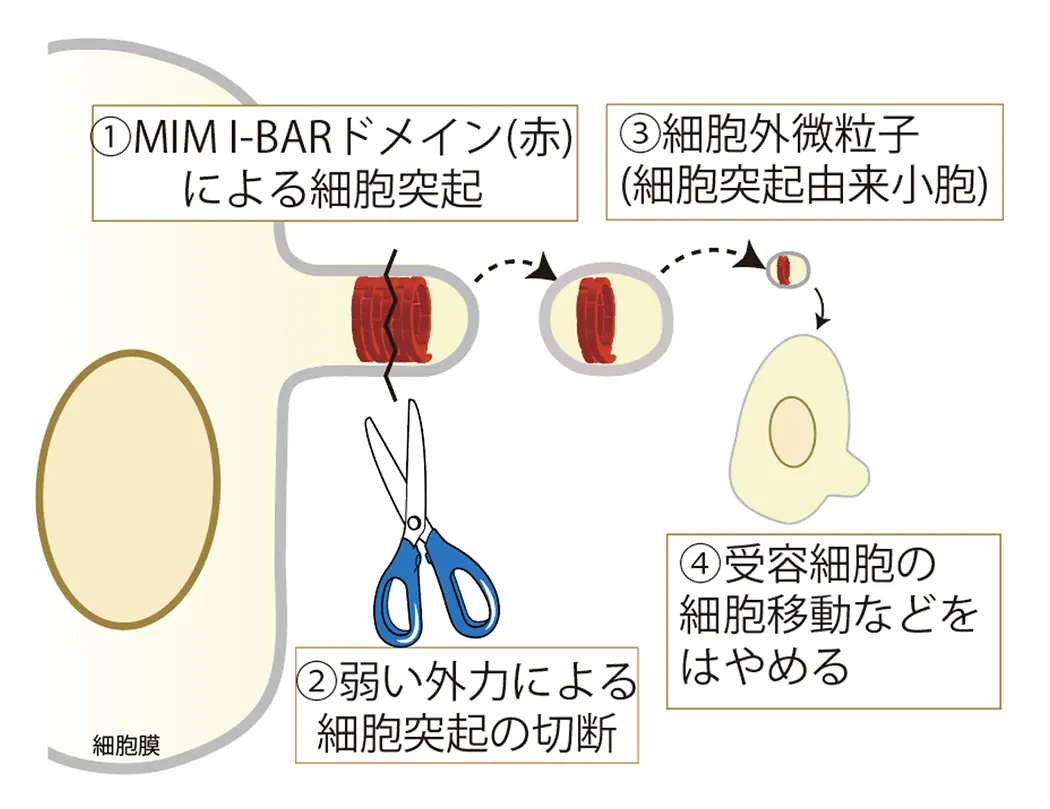
重要な疾患である癌形成やさまざまな疾患において、細胞の形態変化が伴います。細胞の分化、初期化においても同様です。しかし、脂質膜の結合タンパク質がどのように変化、あるいは活性制御を受け、クラスリン被覆小孔、カベオラ、フィロポディア、ラメリポディア、ポドソームなどの細胞小器官の形成に異常が生じ、このような細胞の形態変化が生じるかについてはほとんどわかっていないと言ってよいと思います。バイオサイエンス、バイオナノ理工コースでは、私たちの見出したBARタンパク質などの膜結合性タンパク質や脂質膜の裏打ちとして広く存在するアクチン細胞骨格による細胞構造構築とその細胞内シグナリングにおける役割を明らかにします。膜結合性タンパク質による膜の形態形成の試験管内での再構成を行い、ついで、再構成により得られた知見と細胞機能の相関を調べます。このような研究により、細胞外微粒子(細胞外小胞)の形成機構を見出しました。
主な発表論文・著作
- Hooi Ting Hu, et al., Front. Cell Dev. Biol., 12, 1422227, 2024
- Sim PF., et al., J Biochem, 175, 57-67, 2023
- Wan Mohamad Noor, WNI., et al., Sci Adv., 9, eadf5143, 2023
- Mukherjee, A. et al., Advanced Science (Weinh), 10, e2207368, 2023
- D'Angelo, G. et al., Nature Reviews Molecular Cell Biology, 24, 81-82, 2023
- Nishimura, T. el al., PLoS One., 17, e0271003, 2022
- Osuga, M. et al., Molecular Biology of the Cell, mbcE21010044, 2021
- Shigene, K., et al., Frontiers in Cell and Developmental Biology, 9, 635231, 2021
- Hu, HT., et al., STAR Protocols, 2, 100625, 2021
- Snider, CE. et al., Trends in Cell Biology, 31, 644-655, 2021
- Nishimura, T. et al., Developmental Cell, 56, 842-859, 2021
- Gusmira, A. et al, Journal of Cell Science, 133, jcs246785, 2020
- Kitamata, M. et al., Genes to Cells, 25, 187-196, 2020
- Hanawa-Suetsugu, K., et al., Nature Communications, 10, 4763, 2019
- Kitamata, M. et al., iScience, 17, 101-118, 2019
- Tachikawa, M. et al., Scientific Reports, 7, 7794, 2017
- Senju, Y., et al., Journal of Cell Science, 125, 2766-2780, 2015
- Takahashi, N. et al., Nature Communications, 5, 4994, 2014
- Suetsugu, S. et al., Physiological Reviews, 94, 1219-1248, 2014
- Suetsugu, S. and Gautreau, A., Trends in Cell Biology, 22, 141-150, 2012
- Senju, Y., et al., Journal of Cell Science, 124, 2032-2040, 2011
- Takano, K., et al., EMBO journal, 27, 2817-2828, 2008
- Shimada, A., et al., Cell, 129, 761-772, 2007
(こちらのリンクはすべて外部サイトへ遷移します![]() )
)
 奈良先端科学技術大学院大学
奈良先端科学技術大学院大学