幹細胞工学(栗崎研究室)
-
研究室ホームページ
- https://bsw3.naist.jp/kurisaki/
-
全学オンラインセミナー
2022年度に開催されたBio Discovery Session(全学オンラインセミナー)のアーカイブ動画の一覧です。 - Bio Discovery Session
-
Webメディア
NAIST Edge BIOは、奈良先端科学技術大学院大学 バイオサイエンス領域 の各研究室で取り組んでいる「最先端」の研究プロジェクトや研究成果について、研究者だけではなく受験生や一般の方にも分かりやすく紹介するためのWebメディアです。 - NAIST Edge BIO 第18回
研究・教育の概要
ES細胞やiPS細胞などの多能性幹細胞は、無限の増殖能と体の全ての細胞への分化能を併せ持つ発生初期の幹細胞であり、その強力な多能性と増殖能ゆえに再生医療や創薬などへの応用が期待されています。しかし、これらの幹細胞は発生初期の幹細胞であるがゆえに、目的細胞への分化誘導に長期間を要し、その分化制御には高度な培養技術が必要とされます。一方、最近大人の臓器に存在する微量の幹細胞をオルガノイド(ミニ組織)として培養させ様々な細胞へと分化させる技術が開発されたことで、大人の臓器のしくみを詳しく研究することが可能になってきました。また、正常オルガノイドと疾患オルガノイドを比較することで、シャーレの中で疾患発症のメカニズムを詳しく調べることが可能になってきています。本研究室では、特に胃や肺などの幹細胞の分化制御メカニズムを解析し、組織形成のしくみの解明を進め、さらに疾患モデルや組織再生への応用を目指した研究を行っています。
主な研究テーマ
胃組織の形成と疾患
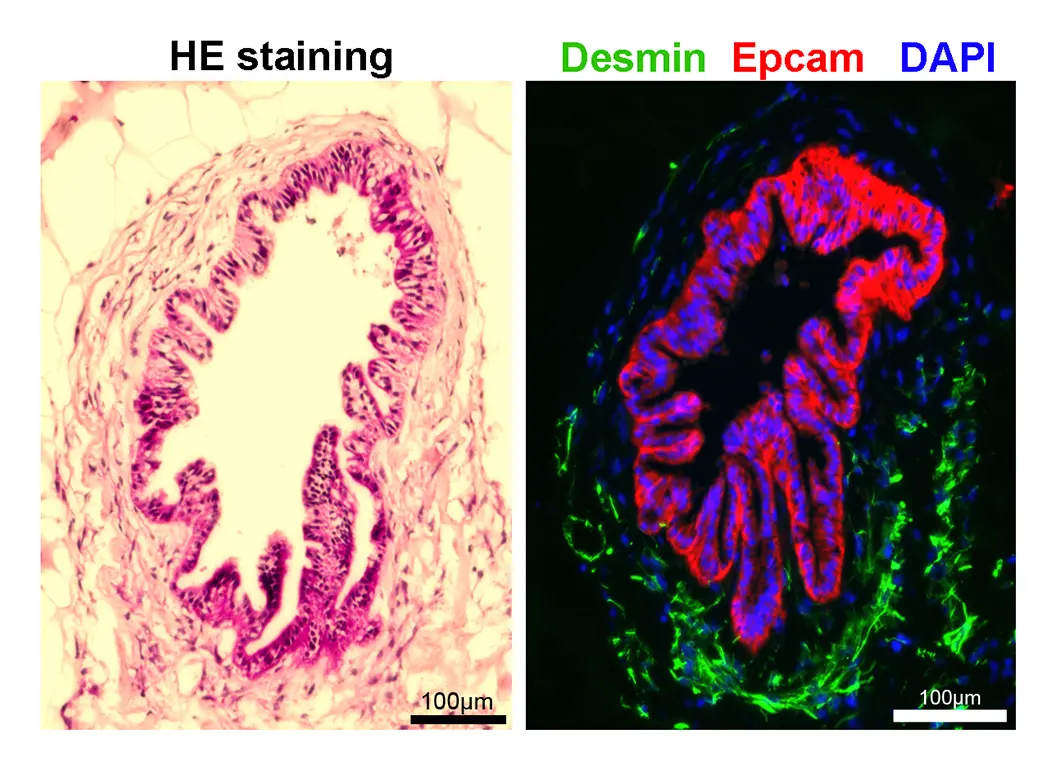
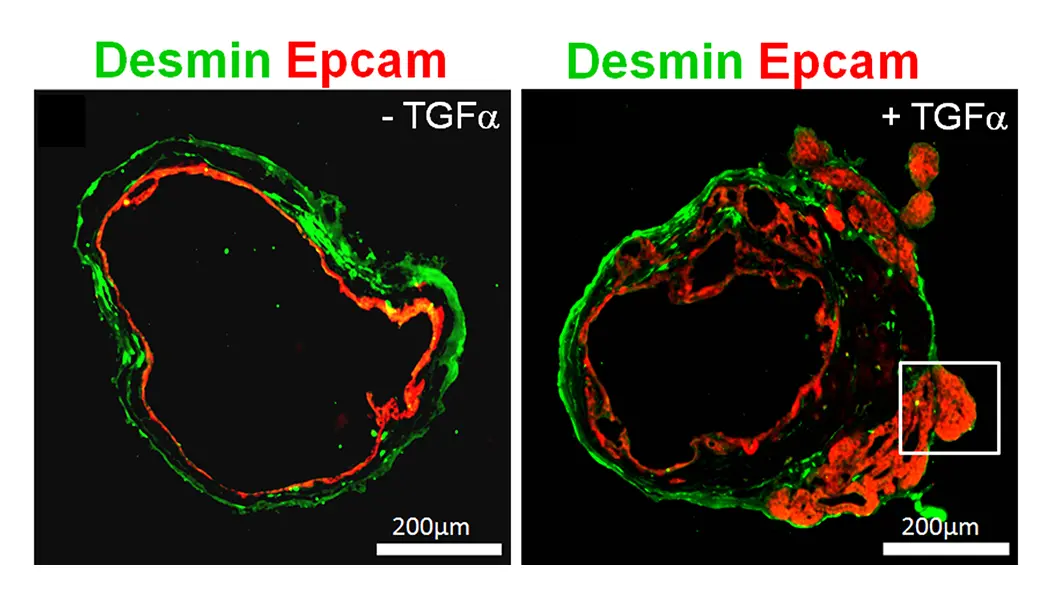
胃は主要な消化器官ですが、その発生のしくみはあまりわかっていません。発生初期の内胚葉から一本の消化管が形成され、胃の予定領域が決定され、さらに成熟分化して胃の内側に数百万個の胃腺と呼ばれる円柱状上皮構造が形成されると考えられていますが、そのメカニズムもあまりわかっていないのです。私たちは、ES細胞から試験管内で胃組織を丸ごとオルガノイドとして作製する方法を報告しました(図1)。また、最近大人の胃の組織幹細胞を増殖させて、再び機能性細胞へと細分化させる方法も開発しました(図3)。この方法は、胃の組織が形成されるしくみを調べるための強力なツールになります。胃がんなどの病気は良い動物モデルが存在しないことから、このような試験管内で作製した胃組織はがんなどの病気の発症機構を調べる上でも有用と考えられます(図2)。また、最近シングルセル解析技術やオルガノイド培養技術を利用して大人の胃の幹細胞から胃粘膜細胞を作り出すしくみや幹細胞を維持するシグナルの解明にも成功しました(図3)。胃の発生のしくみを解析しつつ、オルガノイド分化法を利用して分化のメカニズムを検証するとともに、疾患発症・組織再生機構の解明に取り組んでいます。
肺組織の分化と組織再生
肺は、胃と同じく、発生初期の内胚葉から形成された消化管から、肺の元となる肺芽が出芽し、増殖しながら枝分かれして発生していきます。最近、ヒトiPS細胞から肺の組織細胞を分化させる方法が検討されていますが、私たちも肺前駆細胞や気管支繊毛上皮細胞を分化させる方法の開発を進め、その分化制御のしくみを明らかにするための研究を進めています。
脂肪組織の幹細胞と組織再生
脂肪組織には間葉系幹細胞という線維芽細胞によく似た幹細胞が存在し、生体内で抗炎症作用、血管新生促進作用、再生促進作用などを示すことが知られています。私たちは、脂肪組織に含まれる細胞集団をシングルセルレベルで解析し、その作用メカニズムについて研究を進めています。また、最近発見した抗肥満ホルモンの脂肪組織における作用についても検討を進めています。
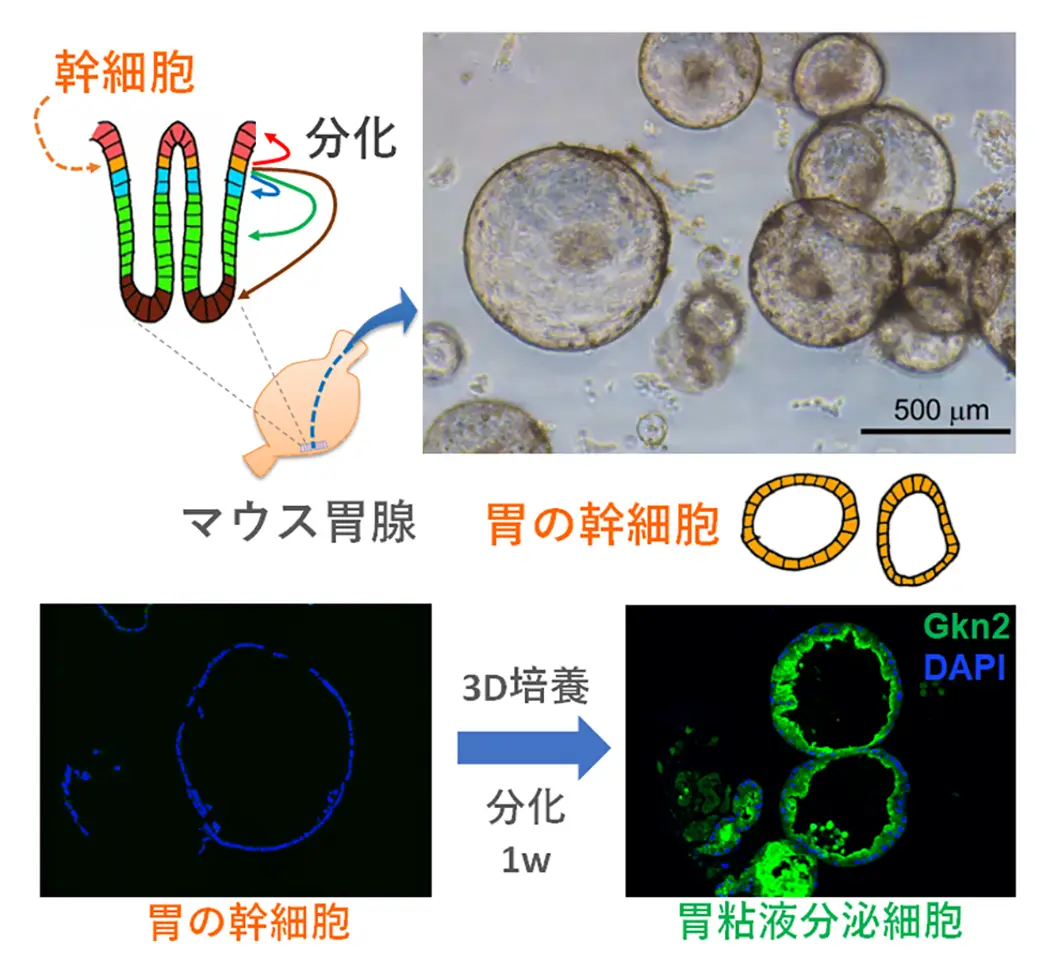
(図3) 胃内壁を構成する胃腺組織から増殖させたマウスの胃の幹細胞オルガノイド。胃腺には、胃酸分泌細胞、消化酵素分泌細胞、粘液分泌細胞や各種内分泌細胞が存在し、これらの機能性上皮細胞は胃腺の幹細胞から分化すると考えられている。実際にこの幹細胞に分化因子を加えて培養すると、1週間で胃粘液分泌細胞へと分化する。
主な発表論文・著作
- Takada H. et al., Nature Comm., 14, 3750, 2023
- Hashimoto O, Cell Rep., 25, 1193-1203, 2018
- Sasagawa Y, Genome Biol. 19, 29, 2018
- Noguchi TK et al., Nature Cell Biology, 17, 984-993, 2015
- Watanabe-Susaki K et al., Stem Cells, 32, 3099-3111, 2014
- Seki Y et al., Proc. Natl. Acad. Sci. U S A, 107, 10926-10931, 2010
(こちらのリンクはすべて外部サイトへ遷移します![]() )
)
 奈良先端科学技術大学院大学
奈良先端科学技術大学院大学