※2024年度まで
原核生物分子遺伝学
教員
 教授
教授
吉田 昭介(兼務)
 准教授
准教授
秋山 昌広
 助教
助教
小林 和夫
研究・教育の概要
私たちの研究室では、(1) 抗菌剤によるヌクレオチド欠乏によって複製阻害や細胞死を生じる時に、ゲノムの不安定性はどのようなプロセスを経て発生するのか、あるいは、(2) 細菌感染症の薬剤治療を妨げる微生物バイオフィルムがどのようなプロセスを経て形成・分解されるのかについて、それぞれ大腸菌と枯草菌を用いて研究を進めています。また、これらの研究課題に興味を持つ若い人達に、研究者や社会人として活躍できる基礎的な力を養えるような教育を全力で行っています。
主な研究テーマ
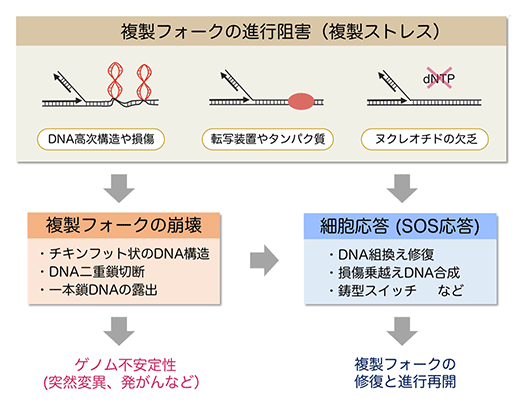
ゲノム不安定性はヌクレオチド不足でどの様に生じるのか?(図1~3)
複製フォークは染色体上を移動しながらDNAを複製します。その進行は、染色体DNAの高次構造や損傷、RNAポリメラーゼやDNA結合タンパク質との衝突、ヌクレオチドの欠乏などの様々な内在性因子によって常に妨げられています。このような複製フォークの進行阻害 (複製ストレス) は、突然変異や染色体異常などのゲノム不安定性を生じて、発がんの主要な原因となります(図1)。
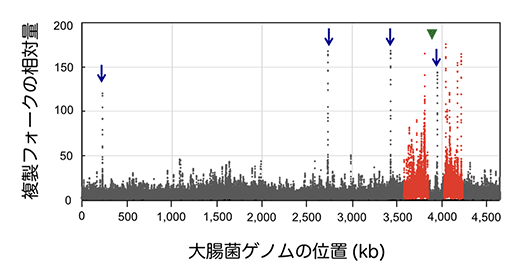
複製フォークはゲノムDNAのどこでどのような原因で停止するのか、また、その停止はどのようにしてゲノム不安定性に繋がるのかを解明するには、染色体上の複製フォークの動態(動き方)を理解することが必要です。しかし、複製フォークで働くDNA複製酵素の基本的な機能が解明されているにも拘わらず、染色体上の複製フォーク動態の全容は微生物でも真核生物でも未解明です。そこで、私たちは、ゲノム構造の単純な大腸菌を用いて、ゲノム上の複製フォークの動態を研究しています(図2)。
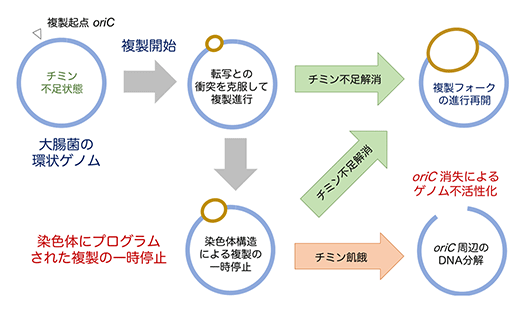
ヌクレオチドの不足は、葉酸 (ビタミンB9) 欠乏時や発がん初期で生じ、ゲノム不安定性を生みます。また、細胞は、ヌクレオチドのチミンを生合成できないチミン欠乏状態になると、複製フォークの進行を阻害されて短時間に劇的な細胞死を起こします(チミン飢餓死)。このチミン飢餓死を引き起こす抗菌剤 (トリメトプリム)や、抗がん剤 (5-FU)は、医療現場で広く使用されています。しかし、その発見から60年以上を経ても、チミン飢餓死で生じるゲノム不安定性や細胞死のメカニズムは明らかにされていません。そこで、私たちは、ヌクレオチド不足状態の大腸菌で、複製フォークの動態を解析しています。これまでに、私たちは、ヌクレオチド不足時に複製フォークを止め、複製をコントロールするようにプログラムされたゲノム領域を発見し、その領域と、チミン飢餓死のゲノム不安定性との関連性を見出しました(図3)。
バイオフィルムはどのようにして形成されるのか?(図4)
環境下ではほとんどの微生物は固体表面に接着した集合体、バイオフィルムを形成し、好ましい環境に定着しています。バイオフィルム中の微生物は、自身が作るポリマーによって保護され、様々な抗菌剤に抵抗性を示します (図4)。そのため、バイオフィルムは、しばしば食品や器具の汚染、感染症の原因となり深刻な問題となっています。しかし、微生物がどのようなシグナルに応答してバイオフィルムを形成するのか、そのメカニズムはいまだ十分に解明されていません。私たちは、遺伝子操作のしやすい枯草菌を用いてバイオフィルム形成機構について解析を進めています。また、バイオフィルムにおける微生物間の競合や物質生産にも興味を持っています。

(図4) 枯草菌のコロニーバイオフィルム
(左)バイオフィルム形成培地では、枯草菌はバイオフィルム形成に伴う複雑な「しわ構造」を持ったコロニーを形成する。(右)ポリマー物質で覆われたコロニー表面は撥水性を示し、水滴(青色)をスポットしても、水滴は拡散しない。
主な発表論文・著作
- T.M. Pham et al., Mol. Microbiology, 90, 584-596, 2013
- K.W. Tan et al., Nucleic Acid Res., 43, 1714-1725, 2015
- M.T. Akiyama et al., Genes to Cells, 21, 907-914, 2016
- K. Kobayashi, J. Bacteriology, 201, e00712-18, 2019
- K. Kobayashi and Y. Ikemoto, PLoS Genetics, 15, e1008232, 2019
- T. Kimura and K. Kobayashi, J. Bacteriology, 202, e00120-20, 2020
(こちらのリンクはすべて外部サイトへ遷移します![]() )
)
 奈良先端科学技術大学院大学
奈良先端科学技術大学院大学