花発生分子遺伝学 (伊藤研究室)


- 教授
- 伊藤 寿朗

- 准教授
- 山口 暢俊

- 助教
- 白川 一

- 和田 七夕子

- 研究室HP
- https://bsw3.naist.jp/ito/
研究・教育の概要
奈良先端大を日本の花発生研究の中心地に!
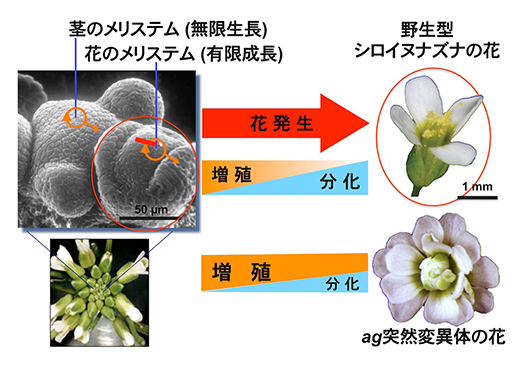
私達は、モデル植物であるシロイヌナズナを用いて、環境に応答した花のかたちづくりの研究をしています。花は観賞用として日常に彩りを与えてくれるだけではなく、穀物や果物を作り出す種子植物の有性生殖のための器官です。花の発生過程においては、花幹細胞の増殖と分化のバランスがダイナミックに変化するため、発生研究のよいモデル系でもあります(図1)。花の形作りの原理を解明することで、植物分野だけではなく、動物の研究者の方々にも注目してもらえるような研究を世界に発信していくことを目指します。日本の花発生研究の中心地となれるよう、学生さん達を大募集中です。
教育においては、バイオ基礎研究を通して問題に真摯に取り組み、解決していく能力を養います。これによりアカデミックのみならず、企業においても応用力、実行力を発揮することのできる人材の育成を行います。
主な研究テーマ
幹細胞の増殖・分化・老化の制御機構
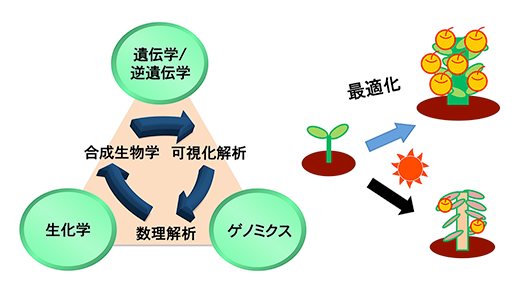
花幹細胞の増殖は多くの転写因子による複数の遺伝学的な経路により抑制されています。それらの転写因子のターゲット遺伝子を同定し、時空間特異的な発現制御機構とターゲット因子の作用機構の研究を行います(図2)。これにより、花幹細胞の増殖抑制経路における植物ホルモンおよびエピジェネティックな制御機構を解明していきます。また、幹細胞から特殊な機能を持つ細胞(虫をおびき寄せる蜜腺細胞や逆に虫の食害を防ぐミロシン細胞など)が分化してくる分子経路を明らかにします。さらに、分化した花器官を作る細胞や茎の幹細胞がどのように、老化して細胞の一生を終えるのかを解明します。可視化解析、数理解析、合成生物学的解析を含めた包括的な解析により、幹細胞の増殖と分化と老化における調和と可塑性、頑強性の機構を分子レベル、細胞レベル、個体レベルで理解することを目指します(図3左)。
環境応答と記憶・忘却の制御機構
植物は、動物とは異なり、環境変動に対して非常に柔軟に応答し、その環境に適応します。高温条件下で活性の高まるヒストン修飾酵素の作用機構などに着目し、植物が環境に対応して、その情報を記憶してメリステムの挙動、分化の様式を変換する機構および、記憶が消去される機構の解明を目指します。さらにこれらの知見を利用して、農業的な視点からより効率的な農作物の開花、結実時期の調節を可能とする基盤技術の構築も目指します(図3右)。
有性生殖におけるエピジェネティック制御機構
アブラナ科植物の自家不和合性に関わる花粉因子には、複雑な優劣性が存在します。また、異なる親同士を交配して得られる雑種第一代は親よりも優れた形質を示すという雑種強勢が見られます。さらに種子では、父方・母方遺伝子の拮抗的な作用機構があり、これらを制御するエピジェネティック機構の解明を目指します。

主な発表論文・著作
- Shirakawa et al., Nature Plants, doi.org/10.1038/s41477-025-01921-1, 2025
- Otsuka et al., Commun. Biol., 8, 108, 2025
- Wang et al., Plant Cell, 36, 5004–5022, 2024
- Wang et al., eLIFE doi.org/10.7554/eLife.100905.1, 2024
- Kobayashi et al., J of Biotech., 392, 103, 2024
- Furuta et al., Nature Commun., 15, 1098, 2024
- Wang et al., Inter J. of Mol Sci., 24, 13297, 2023
- Pelayo et al., Plant Cell, 35, 2821-2847, 2023
- Maruoka et al., Front Plant Sci., 13, 837831, 2022
- Shirakawa et al., Front. Plant Sci., 12, 829541, 2022
- Yamaguchi et al., Nature Commun., 12, 3480, 2021
- Pelayo et al., Current Opinion Plant Biol., 61, 1020009, 2021
- Shirakawa et al., Front. Plant Sci., 12, 634068, 2021
- Wang et al., Front. Plant Sci., 11, 600726, 2020
- Sun et al., Plant Cell, 31, 1488-1505, 2019
- Wu et al., Plant, Cell & Environment, 42, 2198-2214, 2019

 Bio Discovery Session
Bio Discovery Session NAIST Edge BIO
NAIST Edge BIO