mRNA翻訳の効率化
概要
遺伝子の発現において重要な過程の一つがmRNAからタンパク質への翻訳です。mRNAの転写や分解が、蓄積するmRNAの量を決めるのに対し、翻訳はmRNAあたりに作られるタンパク質の量を決めます。そのため、転写に加えて、翻訳の効率も向上させることがタンパク質の生産量を向上させるために重要となります。この翻訳効率は、mRNAの配列によって決定されていますが、特に大きな影響を与えているのは5'UTRと呼ばれる領域です。特に、翻訳を向上させる5'UTR配列は翻訳エンハンサーと呼ばれ、導入遺伝子高発現系の構築に活用されています。当研究室ではこれまでに、植物で非常に強力な翻訳エンハンサーを見出しており、導入遺伝子の発現量を劇的に向上させることに成功しています。現在は、5'UTR配列と翻訳効率との間の関係性を解析することで、目的の有用タンパク質に特化した翻訳エンハンサーを設計する研究を行っています。
非常に強力な翻訳エンハンサーの発見
真核生物の翻訳開始機構は、CAP構造を介したスキャンニングモデルであり、タンパク質合成の速さ(翻訳効率)は、5’非翻訳領域(5’UTR)が仲介する「開始反応」で決まると考えられています(図1)。そのため、同じタンパク質を翻訳する場合(CDS配列が同じ)でも、5'UTRの配列が異なれば、最終的なタンパク質の生産量には大きな差が生じます。特に、翻訳を向上させる5'UTR配列は翻訳エンハンサーと呼ばれ、当研究室では非常に強力な翻訳エンハンサーを見出してきました。まず、発見された強力な翻訳エンハンサーはアルコールデヒドロゲナーゼ遺伝子の5'UTR(ADH-5'UTR)です。シロイヌナズナ由来のAtADH-5'UTRとタバコ由来のNtADH-5'UTRは広い双子葉植物で効果が認められており、既存のpBI系列の発現カセットと比較して約100倍もレポーター遺伝子の発現量を向上させることが一過性発現実験にて示されています。また、イネ由来のOsADH-5’UTRも、広い単子葉植物で、発現上昇効果が認められました。AtADH-5'UTRとOsADH-5’UTRの翻訳エンハンサーは、植物での効率的な発現カセットの一部として商品化されており、タカラバイオ社からpRI201 DNAシリーズとして市販されています。(リンク)
続いて見出されたのが、様々な条件下でも効率的かつ安定的な翻訳が可能な次世代型の翻訳エンハンサーです。mRNAからの翻訳は、細胞が置かれた条件によって変化し、特にストレス環境下や成熟した細胞では、大部分のmRNAにおいて劇的な翻訳抑制が生じることが知られています。有用物質の生産において、培養細胞を用いる場合では、高密度培養時の低酸素や飢餓ストレスを回避することは困難であり、翻訳が抑制されやすい条件下での生産を余儀なくされます。また、植物体を用いる場合では、若い小さな葉よりも大きく成長した葉での生産量が重要ですが、成熟した細胞での翻訳は抑制されがちです。このような条件間での翻訳効率の変化にも5'UTR配列は関わっており、適切な5'UTR配列を用いることで、多くのmRNAからの翻訳が抑制される条件下でも活発な翻訳を行い生産量を向上させることが可能となります。そこで、様々な条件下での翻訳効率を網羅的に評価し、どの条件下でも活発な翻訳が可能な翻訳エンハンサーの探索を行いました。その結果、双子葉植物に対して非常に強力な効果を有するAtCOR47-5'UTRが見出されました。この翻訳エンハンサーは、既存のpBI系列の発現カセットと比較して約180倍、前述したAtADH-5'UTRと比較して約1.8倍もレポーター遺伝子の発現量を向上させることが一過性発現実験にて示されています(図2)。加えて、様々な条件下でも翻訳効率が抑制されないことが形質転換体を用いた発現実験にて確認されています。なお、ADH-5'UTRは、環境ストレス下での翻訳抑制を回避する能力を有していますが、発達した組織では若干抑制される傾向があります。このAtCOR47-5'UTRは、特許第6607616号として特許化されています。注意点として、AtCOR47-5'UTRは単子葉植物のイネでは全く機能せず、逆に翻訳を抑制します。当研究室では、単子葉植物でも環境ストレス下での翻訳効率の網羅的な解析を行っており、単子葉で機能しストレスによる翻訳抑制を回避できる翻訳エンハンサー配列を多数取得しています。これらの配列は、WO2019/198724として国際特許出願されています。
こうして得られた翻訳エンハンサーは、配列の一部に改良を加えていますが基本的には植物のmRNAが元々有している5'UTRの配列です。現在は、5'UTR配列と翻訳効率の間の関係性を解き明かすことで、より効率的な翻訳を可能とする全く新しい配列を設計する研究を行っています。詳細は下記の「目的の有用遺伝子に特化した翻訳エンハンサーの設計」をご覧ください。
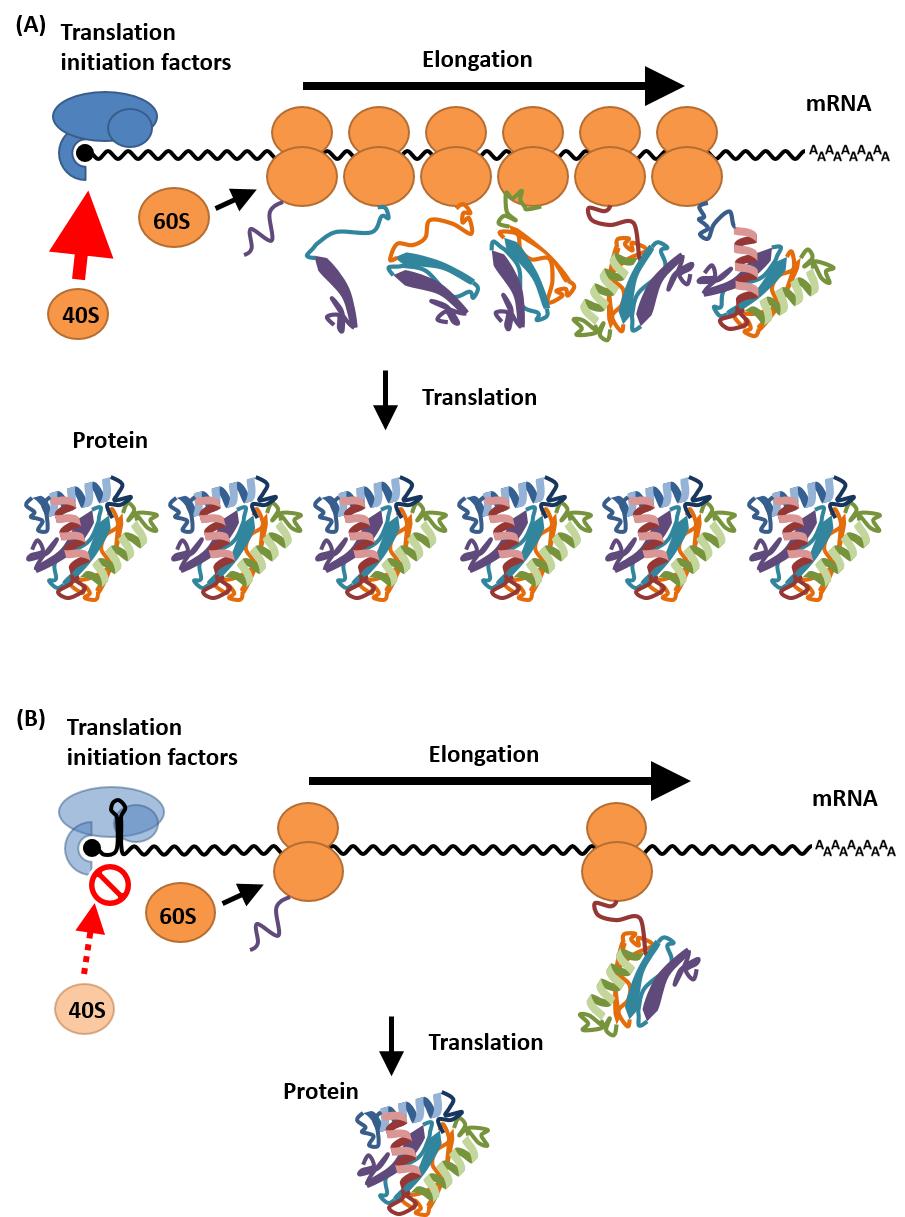
図1 5'UTRの配列と翻訳効率
(A)翻訳効率の決定には翻訳開始因子群による40Sリボソーム(正確には43S複合体)のリクルートが重要となります。高効率でのリクルートが可能な5'末端配列(主に5'UTRに相当)を有している場合、mRNAはポリソームを形成し活発な翻訳が行われ、単位mRNAあたりに多くのタンパク質を合成することができます。(B)5'UTR領域の特定の領域に強固な二次構造を有する場合などでは、リボソームのリクルートが阻害されます。この場合、リボソームは低頻度でしかリクルートされず、単位mRNAあたりのタンパク質の合成量は低くなってしまいます。
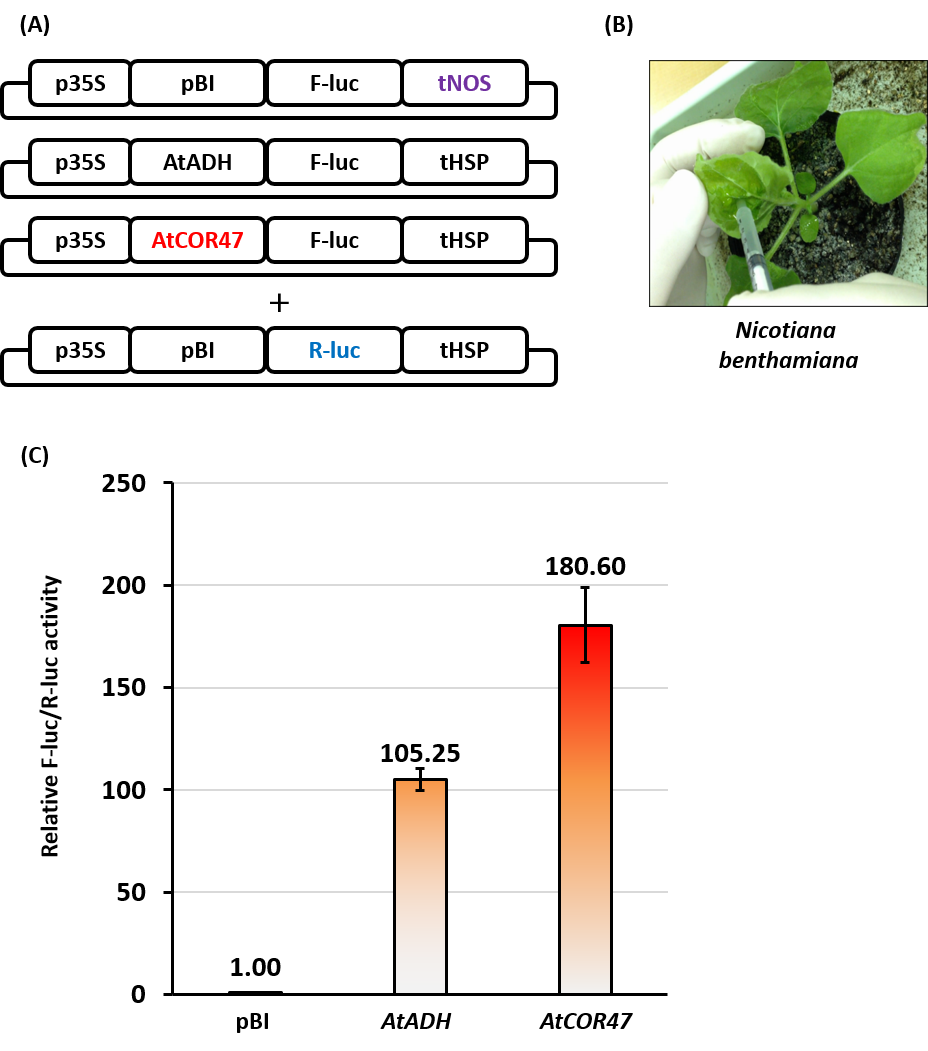
図2 翻訳エンハンサーによる発現量の向上
(A)翻訳エンハンサー能力がレポーター遺伝子(F-luc)の発現能力に与える影響を評価するために構築した発現ベクターの模式図。ベクター由来の5'UTR領域とNOSターミネーターを有し、植物で一般的に使用されるpBI系の発現ベクターと、当研究室で見出されたAtADH遺伝子とAtCOR47遺伝子由来の翻訳エンハンサーおよびHSPターミネーターを組み合わせた発現ベクターを使用しています。実験上の導入効率等の差を補正するために、各測定には、補正用のR-luc遺伝子も共導入しています。(B)構築した発現ベクターを、タバコ葉にアグロインフィルトレーションによって一過的に導入している様子。(C)F-lucタンパク質の活性値を測定した結果。既存の発現ベクターと比較し、AtADH遺伝子由来の翻訳エンハンサーを使用した場合では100倍以上、AtCOR47遺伝子由来の翻訳エンハンサーを使用した場合では180倍以上の発現能力の向上に成功しています。
目的の有用遺伝子に特化した翻訳エンハンサーの設計
mRNAの翻訳効率は、mRNAの配列、特に5'UTRの配列によって決定されています。このような翻訳効率と配列の関係性を解き明かすことができれば、解き明かした関係性に基づいて、より効率的な翻訳を可能とする全く新しい配列を設計することが可能になります。加えて、当研究室で行われた、翻訳効率の決定に重要なmRNAの配列的特徴の解析から、5'UTRの配列はそれ単体で翻訳効率に影響を及ぼすだけではなく、5'UTRとCDSの間の二次構造など、CDSの配列とともに翻訳効率に影響を及ぼしていることが分かりました。このように5'UTRとCDSの間で形成される特徴が翻訳効率に関わっていることから、目的の有用タンパク質をコードするCDS配列によって最適な翻訳エンハンサー配列は異なっていることが想定されました。そこで、現在は目的の遺伝子に特化した全く新しい翻訳エンハンサー配列を設計する研究を行っています(図3)。具体的には、当研究室で取得した膨大な翻訳効率とmRNAの配列情報から、翻訳効率とmRNA配列との関係性を機械学習法を用いて学習させます。学習させた機械学習モデルは、mRNA配列から翻訳効率を予測することを可能とします。後は、「ある配列を翻訳エンハンサーとして目的遺伝子のCDS配列に連結した場合」の翻訳効率を予測し、より予測翻訳効率が高くなるように配列を置換していけば、その遺伝子に特化した配列を取得することができます。翻訳効率の予測には、複数の機械学習モデルをスタッキングしたものを用い、配列の置換には、最適化アルゴリズムの一つである分散並列遺伝的アルゴリズムを使用しています。既に、最適翻訳エンハンサー配列の設計システムの基本は完成しており、数年の時間をかけて発見した既存の翻訳エンハンサーと同等以上の配列を、数時間で設計することに成功しています。
現在は、機械学習モデルによる予測精度を向上させるために、より高精度かつ高解像度の翻訳効率の評価や、より詳細なmRNA配列の同定などの学習データの改良を行っています。
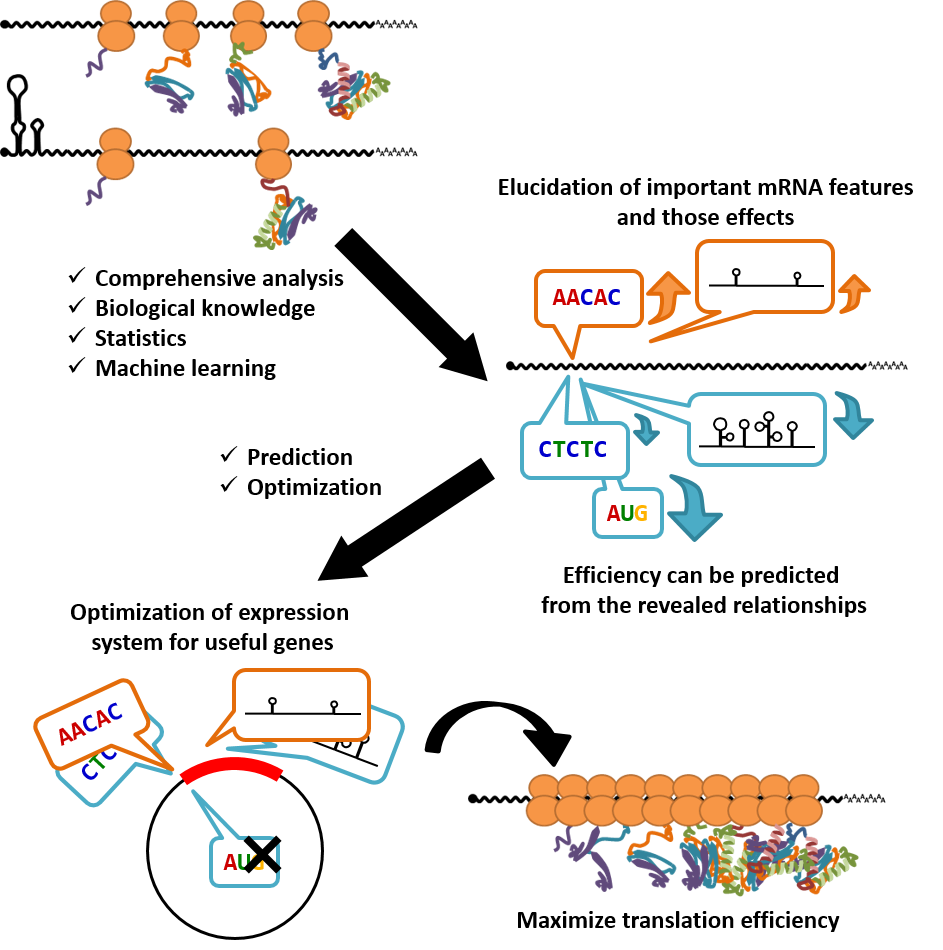
図3 翻訳エンハンサーの設計の模式図
mRNAの翻訳状態を網羅的に解析し、データ解析によって得られた知見と、既存の生物学的知見、統計学、機械学習等を用いて翻訳の効率を決定する要因をモデル化します。これによって、mRNA配列から翻訳効率を予測することが可能となります。そのため、in silicoにて発現ベクターの配列の改変と翻訳効率の予測を繰り返すことによって、翻訳効率を極大化するような配列を設計することが可能となります。
翻訳伸長反応の停止・停滞を回避するコドン最適化
mRNAの翻訳効率には、リボソームのリクルート効率が重要であることは前述した通りです。そしてリボソームのリクルート効率に続いて重要となるのがリボソームによる翻訳伸長反応の速度であると考えられています。例えば、対応するtRNA量が豊富に存在するコドンがCDS内に多く存在する場合、リボソームの翻訳伸長速度が早い (翻訳効率が高い)こと(図4A)、その反対に対応するtRNA量が少ないコドンが多い場合は、リボソームの翻訳伸長速度は遅くなることが(図4B)、酵母において報告されていいます。また、コーディング領域のmRNAの二次構造が、翻訳効率に影響を与えることが出芽酵母において報告されています。このように、同じアミノ酸配列でも各アミノ酸に対応するどのコドンを用いるのかによって翻訳の効率は変化します。加えて、翻訳伸長速度とmRNAの安定性には強い関係性があることが報告されており、伸長速度が速いほど安定的であると考えられています。そのため、翻訳伸長反応の停止・停滞を回避するようにコドンを最適化することによって、翻訳の効率化とmRNAの安定化の両方の効果を期待することができます。当研究室では、翻訳伸長反応の停止・停滞を解析することができるリボソームフットプリント法を用いて、植物mRNA上のどの位置で翻訳伸長反応の停止・停滞が生じているのかを網羅的に評価しています。
現在は、当研究室で得たデータや公共データベースに登録されているデータを用いて、どのようなコドンおよび配列的な特徴が翻訳伸長反応の停止・停滞に関わっているのかの解析を行っています。
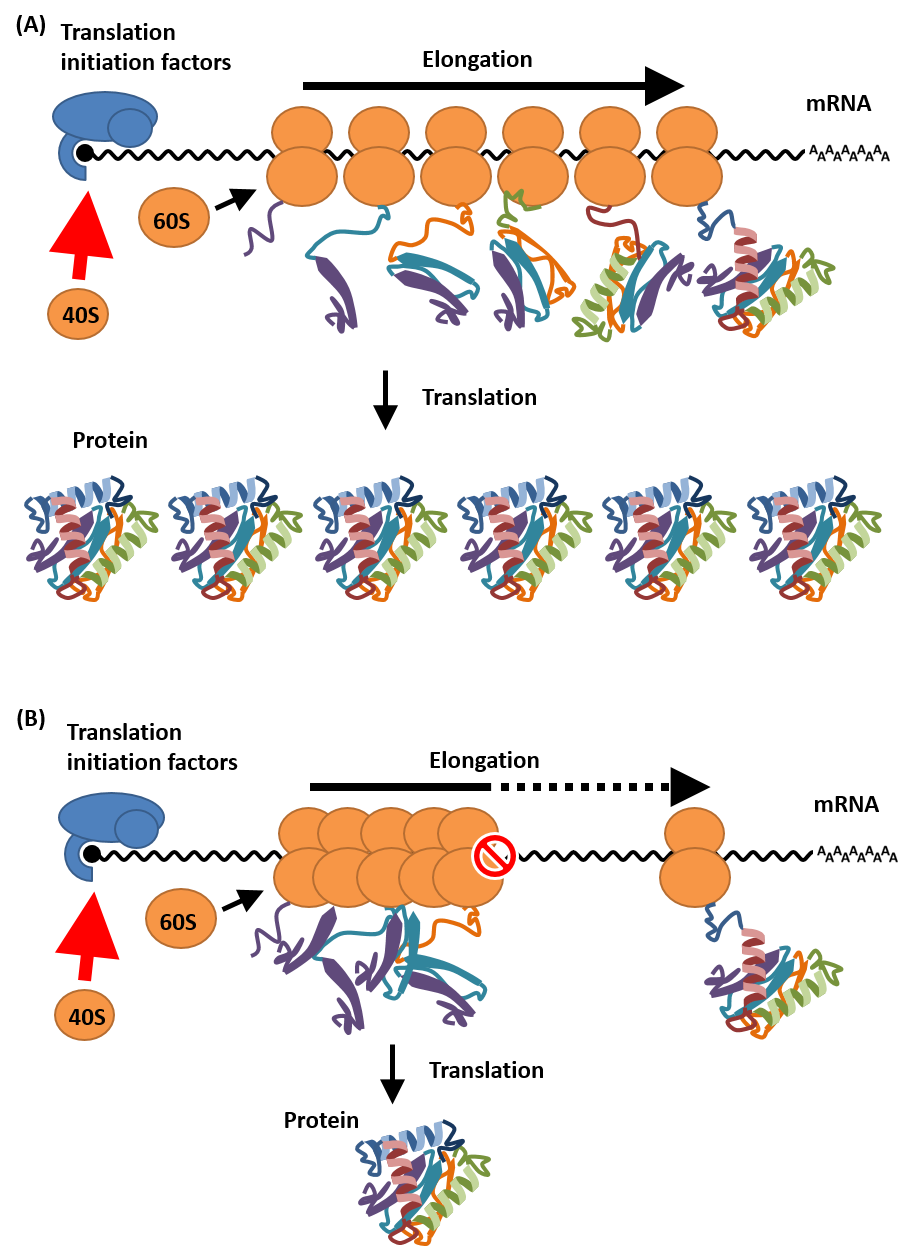
図4 翻訳伸長反応の停止・停滞と翻訳効率
(A)リボソームが効率的にリクルートされ、スムーズな翻訳伸長反応が起こっている場合の模式図。(B)リボソームは効率的にリクルートされているものの、特定の位置で翻訳伸長反応の停止が生じている場合の模式図。リボソームが多くリクルートされた場合でも、伸長反応の停止が生じる場合では、そこでリボソームの渋滞が生じることによって最終的な翻訳効率が低下します。