次世代シーケンサーを用いた基盤情報の取得

概要
近年の生物の研究を支える重要な技術の一つが次世代シーケンサーです。この次世代シーケンサーは、大量の塩基配列を高速で読むことが可能な機械であり、そこから得られる配列情報は遺伝子の研究をする上で必須の情報となっています。この次世代シーケンサーには、その特性や目的によって複数の種類が存在しています。その中でも、Nanoporeシーケンサーに代表されるロングリードシーケンサーは、最も先進的なシーケンサーであり、従来法に存在していた様々な問題を一挙に解決できると期待されています。一方で、その技術は未だ発展途上であり、ハード面でもソフト面でも多くの課題を抱えています。そこで当研究室では、ロングリードシーケンサーによって得られるデータを処理、解析するツールの開発を行っています。
ロングリードシーケンサーを用いた全長mRNA配列の取得
遺伝子から転写されるmRNAの配列とその蓄積量を知ることは、遺伝子発現を理解し生命現象を明らかにする上で必須のステップです。加えて、近年の研究によって、生物は一つの遺伝子からスプライシングパターンや転写開始点、ポリA付加部位などが異なる様々なバリアントを転写しており、それらバリアントの間では翻訳されるタンパク質の機能や翻訳効率、mRNAの安定性などが異なっていることが分かってきています。そのため、正確なmRNAの配列を知ることの重要性が認識されるようになってきました。一方で、一般的なショートリードシーケンサーを用いてmRNA配列を同定する場合、mRNA配列の全長をそのまま読むことができないため断片化の過程が必要となります。この断片化の過程によって、通常のRNA-seqなどではmRNAの5'および3'末端の位置や、スプライシングパターンを正確に同定することが困難となっています。この問題に対応するため、ショートリードシーケンサーを用いた解析では、5'末端の解析だけに特化したCap Analysis of Gene Expression(CAGE)法のように特定の目的に特化した様々な手法が編み出されています。一方で、近年登場したロングリードシーケンサーのNanoporeシーケンサーを用いれば、断片化をせずにmRNAの配列を5'から3'末端までの全長を同定することが可能となっています。これは従来法では別々に行っていた特定部位の詳細解析を同時に行えることを意味し、これまでは推定に留まっていた全長のmRNA配列を正確に同定することが可能です。このようにNanoporeシーケンサーは、非常に多くの可能性を有した機械であると言えます。しかし、シーケンス精度の低さや、不完全長のリードの混入などのハード面での問題や、解析ツールの不足などソフト面での問題を抱えています。そこで、本研究室では、データ処理によって信頼できる情報のみを選抜し、同定した全長のmRNA配列から、バリアントレベルで遺伝子の発現を解析できるツールの開発を行い、実際に複数の生物種の様々な条件下でデータの取得を行っています(図1)。こうして得られた情報は、当研究室で行っている導入遺伝子高発現系の配列最適化での学習データなど、配列情報の正確性が特に求められる高度なデータ解析に特に有用です。
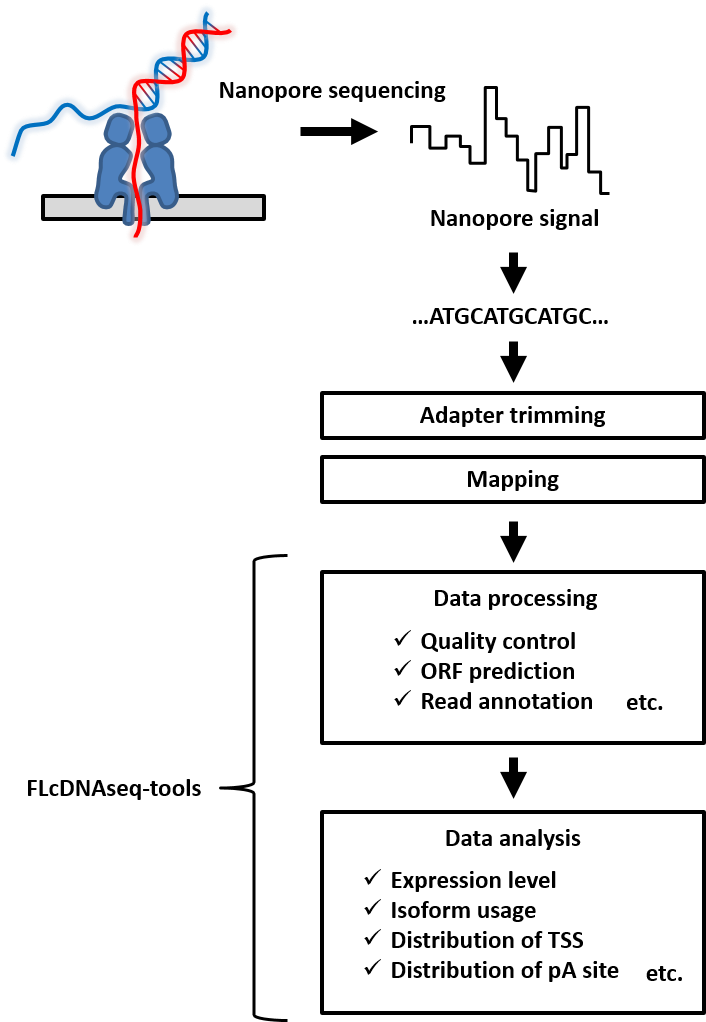
図1 開発中の全長mRNA配列の解析ツールの概要
Nanoporeシーケンサーのマッピングデータについて、品質管理やORF予測、リードアノテーションなどのデータ処理と、バリアントレベルでの発現レベルやアイソフォームの比率、転写開始点やポリA付加部位の分布などのデータ解析を行えるツールを開発中です。