研究成果の紹介
遺伝子の転写開始点の検出法 TSS-seq2 を開発
――メッセンジャーRNA の 5’末端を高い特異性で検出――
遺伝子の転写開始点の検出法 TSS-seq2 を開発
――メッセンジャーRNA の 5’末端を高い特異性で検出――
【 発表のポイント】
◆ゲノム DNA から遺伝子を読み取る開始位置である転写開始点(TSS)を検出する TSS-seq2 法を開発し、4 種類の植物の転写開始点の情報を収集しました。
◆既存の方法よりも高い特異性で転写開始点が検出でき、少量の RNA でも実施できることを示しました。
◆今回開発した方法は既に応用が開始されており、希少な細胞種や微小な組織などの少量のサンプルでの mRNA の正確な構造の同定や遺伝子の制御の研究への貢献が期待されます。
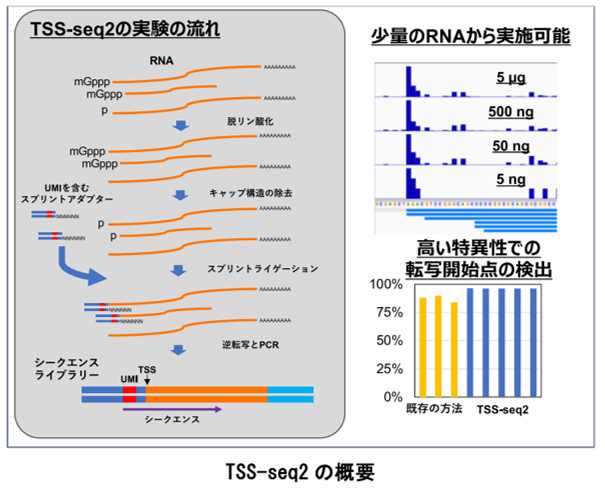
【概要】
東京大学大学院新領域創成科学研究科の関真秀特任准教授と鈴木穣教授、京都大学大学院理 学研究科の松下智直教授と野田口理孝教授(兼:名古屋大学生物機能利用開発研究センター 特任教授)、奈良先端科学技術大学院大学先端科学技術研究科の吉田聡子教授、筑波大学生命環境系の壽崎拓哉准教授、名古屋大学生物機能開発利用研究センターの黒谷賢一特任准教授らの研究グループは、ゲノム DNA から遺伝子を読み取る開始位置である転写開始点(TSS)を網羅的に決定する TSS-seq2 法を開発しました。さらに、コシオガマ、ベンサミアナタバコ、ミヤコグサ、ハクサンハタザオの 4 種類の植物について、TSS 情報の収集を行いました。
TSS の決定は、RNA の正確な構造や、TSS の近辺に存在して遺伝子の機能を調節する重要な領域であるプロモーター領域を同定するために重要です。正確性の高い転写開始点検出法は、必要な RNA の量が多く、プロトコルが複雑であるなど、簡単には実施できない手法が主流でした。今回、先行研究により開発された TSS-seq(注 1)を改良・簡略化することで、TSS-seq2 を開発しました。TSS-seq2 は既存の方法よりも特異的に転写開始点を検出でき、5 ナノグラムと少量の RNA からでも実施できます(図 1)。
今回開発した方法は、様々な生物種や組織での mRNA の正確な構造の同定や遺伝子の制御の研究、特に、希少な細胞種や微小な組織などの少量のサンプルの解析への応用が期待されます。また、今回収集した植物の TSS 情報は、これらの植物種の研究の基盤データとなることが期待されます。
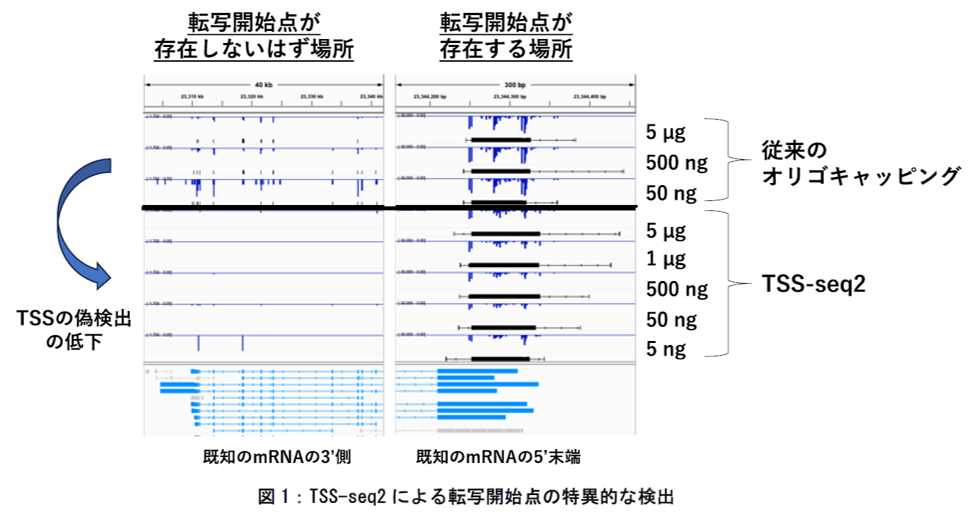
【発表内容】
ゲノム DNA から mRNA が合成される転写は、転写開始点(TSS)と呼ばれる位置から開始され ます。そのため、mRNA の 5’末端の配列を読み取ることで TSS を決定することができます。TSSを決定することで、mRNA の配列の正確な構造を明らかにすることができます。また、TSS の近辺はプロモーター領域と呼ばれる、遺伝子の機能を調節する重要な領域であるため、TSS を検出することでプロモーターを同定することができます。
TSS-seq を含む TSS を正確に決定する主な方法では、数十から数マイクログラムの大量の RNA が必要で、数十時間かかるなど手順が複雑で簡単には実施できないなどの問題点が存在していました。そこで、2009 年に鈴木穣教授らによって開発された TSS-seq 法を改良して、TSS-seq2 法を開発しました。具体的には、オリゴキャッピング法(注 2)に用いられる RNA 同士をつなげるライゲーション(注 3)の方法の変更や PCR(注 4)によって生まれる分子の数の偏りを補正するための RNA の配列を導入するなどの変更を行いました。これにより、TSS-seq2 は 7−8 時間程度の比較的短い時間で実施ができるようになり、最少 5ng と少ない RNA 量からでも実施できることを示しました。さらに、TSS の検出の特異性を TSS-seq を含む既存の方法と比較したところ、TSS-seq2 が最も特異的に TSS を検出できることを示しました。
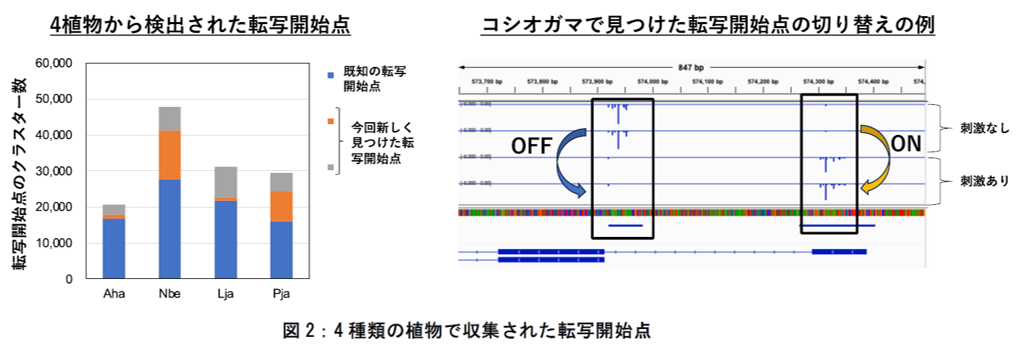
さらに、これまでほとんど TSS の解析がされていなかった 4 種類の植物種(コシオガマ、ベンサミアナタバコ、ミヤコグサ、ハクサンハタザオ)に TSS-seq2 を適用しました(図 2)。すると、既存の遺伝子情報に含まれない約 4,000-20,000 カ所の転写開始点を新たに検出することができました。さらに、他の植物に寄生する性質を持つコシオガマについて、寄生の際に重要な吸器の形成を誘導する刺激を与えたところ、転写開始点の使用頻度の変化を示す遺伝子を検出することができました。
今回開発した TSS-seq2 は、希少な細胞種や微小な組織などの少量サンプルでの TSS 解析への応用が期待されます。また、今回収集した植物の TSS 情報は、遺伝子構造の同定や転写制御の解析への基盤データとなることが期待されます。すでに TSS-seq2 は、文部科学省科学研究費助成事業 学術変革領域「不均一環境と植物」及び「先進ゲノム支援」の支援活動を通じて、植物を始めとした様々な生物種に応用され始めています。今後、様々な生物種や環境適応における転写開始点制御の役割を明らかにするための研究への寄与が期待されます。
【発表者・研究者等情報】
東京大学大学院新領域創成科学研究科
関 真秀 特任准教授
鈴木 穣 教授
京都大学大学院理学研究科
松下 智直 教授
野田口 理孝 教授 兼:名古屋大学生物機能利用開発研究センター 特任教授
奈良先端科学技術大学院大学先端科学技術研究科
吉田 聡子 教授
筑波大学生命環境系
壽崎 拓哉 准教授
名古屋大学生物機能開発利用研究センター
黒谷 賢一 特任准教授
【論文情報】
雑誌名:Nucleic Acids Research
題 名 :An improved method for the highly specific detection of transcription start sites
著者名:Masahide Seki*, Yuta Kuze, Xiang Zhang, Ken-ichi Kurotani, Michitaka Notaguchi, Haruki Nishio, Hiroshi Kudoh, Takuya Suzaki, Satoko Yoshida, Sumio Sugano, Tomonao Matsushita, Yutaka Suzuki*
DOI:10.1093/nar/gkad1116
【研究助成】
本研究は、科研費「不均一環境変動に対する植物のレジリエンスを支える多層的情報統御の分子機構(課題番号:JP20H05905)」、「植物の不均一環境変動へのレジリエンスを支える転写開始点制御機構(課題番号:JP20H05906)」、「不均一土壌環境に応答した寄生植物の感染統御機構(課題番号:JP20H05909)」、「複合的な不均一環境における根粒共生を介した窒素栄養獲得の統御機構(課題番号:JP20H05908)」、「変動環境下での頑健な応答を支える長期クロマチン記憶(課題番号: JP21H04977 )」、「不均一な環境下で全身移行する mRNA に関する研究( 課題番号: JP21H05657)」、「不規則な環境変動下における花成抑制遺伝子 FLC の頑健な制御(課題番号: JP21H05659)」、「先進ゲノム解析研究推進プラットフォーム(課題番号:JP22H04925)」の支援により実施されました。
【用語解説】
(注 1)TSS-seq
オリゴキャッピング法(注 2)を行った mRNA を DNA に変換して、大量の DNA の配列を一度に解読できるハイスループットシークエンサーで読み取ることで転写開始点を網羅的に決定する方法。
(注 2)オリゴキャッピング法
mRNA の 5’末端に存在するキャップ構造を RNA オリゴに置き換える方法。
(注 3)ライゲーション
DNA や RNA などの核酸同士を結合させる反応。TSS-seq で採用されていた 1 本の鎖の RNA 同士のライゲーションは、比較的効率が低く、塩基配列による反応の偏りがあるとされていました。TSS-seq2 では RNA 同士のライゲーション反応をスプリントライゲーションとよばれる方法に変更することで、反応の高効率化や偏りの低減を実現しました。
(注 4)PCR
ポリメラーゼ連鎖反応法とよばれる DNA をコピーして増やす方法。PCR で増やされた DNA はその配列によって、増え方に偏りがあります。そのため、コピー元の DNA の数を正確に数えるためには、元の DNA ごとに別々の配列を予め入れておくなどの方法が取られます。
【植物共生学研究室】
研究室紹介ページ:https://bsw3.naist.jp/courses/courses113.html
研究室ホームページ:https://bsw3.naist.jp/yoshida/
(2023年12月04日掲載)
