酵母のストレス耐性機構の解明と産業酵母の育種への応用
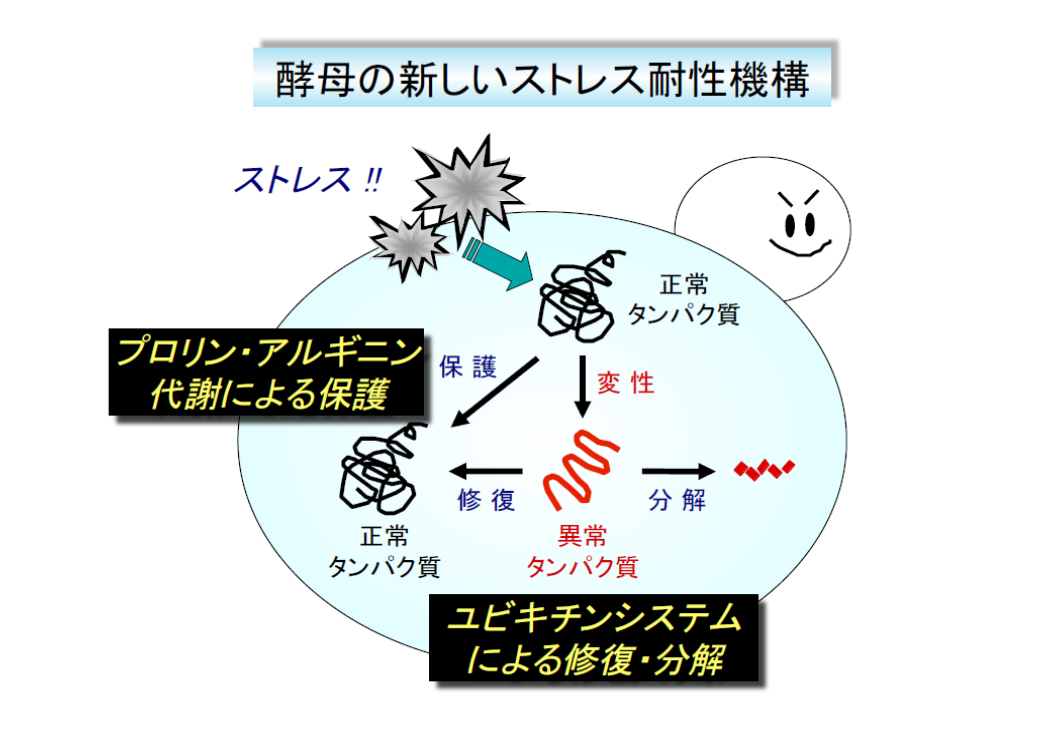
微生物は様々な環境ストレスに応答し、細胞内の遺伝子発現・タンパク質活性・代謝バランス等を巧妙に調節することで、恒常性を維持しています。私たちは高等生物のモデル生物として重要な酵母Saccharomyces cerevisiaeを対象とし、ストレスに対する細胞の応答・適応・耐性機構を解析しています。また、酵母はパン類・酒類・発酵食品(醤油、味噌)・バイオ燃料などの製造に用いられ、8兆円もの巨大産業を支える微生物です。
酵母の発酵生産過程はストレス環境であり、細胞は高濃度エタノール、高温、冷凍、乾燥、高浸透圧、偏栄養など多様なストレスに曝されています。このようなストレスを連続的・複合的に受けると、多くの細胞内タンパク質は変性し、正常な構造や機能を失った「異常タンパク質」になります。また、ミトコンドリア膜の損傷や抗酸化酵素の失活などによって細胞内に活性酸素種(ROS)が蓄積し、酵母の有用機能(エタノール、炭酸ガス、味・風味成分の生成)が制限されます。したがって、発酵食品やバイオエタノールの生産性向上には、酵母に優れたストレス耐性を付与することが重要です。
私たちは、独自に見出した酵母のストレス耐性機構について、以下のようなテーマに取り組み、得られた研究成果を産業酵母の育種に応用することをめざしています。
1. プロリン・アルギニン代謝を介したストレス耐性機構
1) プロリンの代謝制御機構と生理的役割
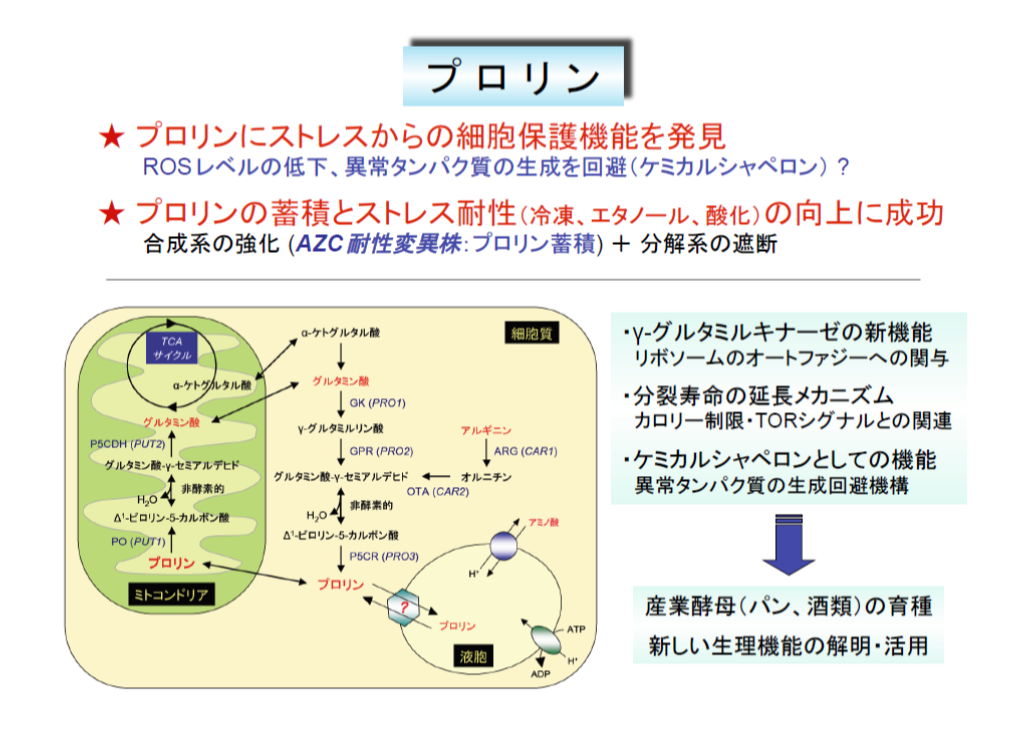
私たちは以前に、アミノ酸のプロリン(Pro)が冷凍後の酵母細胞の生存率低下を抑えることを見出しました。Proには、浸透圧の調節、タンパク質や細胞膜の安定化、ヒドロキシラジカルの消去、核酸のTm値低下、氷結晶形成や脱水の防止などが報告されていますが、細胞内での役割はほとんど分かっていません。
多くの細菌や植物では、乾燥や塩ストレスに応答してProを蓄積しますが、酵母はストレス時にPro合成を誘導せず、トレハロースやグリセロールを蓄積します。そこで、Proと競合して新生タンパク質に取込まれ、異常タンパク質を生成させるProアナログ(アゼチジン-2-カルボン酸;AZC)を用い、細胞内にProを蓄積する変異株を分離しました。AZCに耐性を示す変異株の中から、Pro蓄積株を分離し、解析した結果、γ-グルタミルキナーゼ(Pro1)の遺伝子に変異が入ると(Asp154Asn, Ile150Thrなど)、Proによるフィードバック阻害感受性が低下し、Proを過剰合成することが判明しました。また、Proオキシダーゼ(Put1)の遺伝子破壊株で変異型Pro1を発現させると、Pro含量の増加と冷凍耐性の向上が見られました。Pro蓄積株は冷凍以外に乾燥、酸化、浸透圧、エタノールなどのストレス下でも生存率が高く、Proの細胞保護効果を確認できました。さらに、産業酵母(パン酵母、清酒酵母)で変異型Pro1を発現させると、Proが細胞内に蓄積し、高糖生地や冷凍生地での発酵力、エタノール生産性などが向上しました。最近では、実用化を目指したパン酵母の育種にも取り組んでおり、AZC耐性変異株の中から、Pro1遺伝子に新規な変異が入り、親株に比べてPro含量および冷凍生地・高糖生地での発酵力が向上した株を取得し、Proがパン酵母の育種に有効であることを実証しました(日本農芸化学会2017年度大会トピックス賞)。
一方、Proの生理的役割についても解析し、ストレス下における細胞内ROSレベルの制御、液胞への局在の重要性、リボソームの選択的オートファジー(リボファジー)への関与、細胞寿命の延長効果などを見出しています(東京工業大学、長浜バイオ大学との共同研究)。ProはミトコンドリアのPut1で酸化的に分解され、エネルギー源や窒素源となります。また、高等生物ではProとその代謝中間体P5Cが細胞内レドックスを制御し、エネルギー産生やアポトーシスなどに関与しています。これらの現象には、細胞質で合成されたProのミトコンドリアへの輸送が必要であるため、ミトコンドリアのProトランスポーターの同定を試みています。
<発表論文>
- A. Nishimura, Y. Yoshikawa, K. Ichikawa, T. Takemoto, R. Tanahashi and H. Takagi: Longevity regulation by proline oxidation in yeast. Microorganisms, 9(8), 1650 (2021).
- 西村 明, 高崎友里恵, 高木博史: 酵母におけるプロリンの新しい生理機能と代謝調節機構. 生化学, 93, 203-211 (2021).
- A. Nishimura, R. Tanahashi and H. Takagi: The yeast α-arrestin Art3 is a key regulator for arginine-induced endocytosis of the high-affinity proline transporter Put4. Biochem. Biophys. Res. Commun., 416-421 (2020)
- N. Murakami, A. Kotaka, S. Isogai, K. Ashida, A. Nishimura, K. Matsumura, Y. Hata, H. Ishida and H. Takagi: Effects of a novel variant of the yeast γ-glutamyl kinase Pro1 on its enzymatic activity and sake brewing. J. Ind. Microbiol. Biotechnol., 47, 715-723 (2020). DOI: 10.1007/s10295-020-02297-1
- A. Nishimura, T. Tanikawa and H. Takagi: Inhibitory effect of arginine on proline utilization in Saccharomyces cerevisiae. Yeast, 37, 531-540 (2020). DOI: 10.1002/yea.3504
- Y. Mukai, Y. Kamei, X. Liu, S. Jiang, Y. Sugimoto, N. S. M. Nanyan, D. Watanabe, H. Takagi: Proline metabolism regulates replicative lifespan in the yeast Saccharomyces cerevisiae. Microbial Cell, 6, 482-490 (2019).
- 高木博史: 酵母に見出したプロリン/アルギニン代謝に基づく冷凍・乾燥ストレス耐性機構とパン酵母の育種への応用. 低温生物工学会誌, 63, 11-20 (2017).
- A. Tsolmonbaatar, K. Hashida, Y. Sugimoto, D. Watanabe, S. Furukawa and H. Takagi: Isolation of baker’s yeast mutants with proline accumulation that showed enhanced tolerance to baking-associated stresses. Int. J. Food Microbiol., 238, 233-240 (2016). 「日本農芸化学会2017年度大会トピックス賞」受賞
- I. Nishida, D. Watanabe and H. Takagi: Putative mitochondrial α-ketoglutarate-dependent dioxygenase Fmp12 controls utilization of proline as an energy source in Saccharomyces cerevisiae. Microbial Cell, 3, 391-397 (2016).
- Y. Tatehashi, D. Watanabe and H. Takagi: g-Glutamyl kinase is involved in selective autophagy of ribosomes in Saccharomyces cerevisiae. FEBS Lett., 590, 2906-2914 (2016).
- I. Nishida*, D. Watanabe*, A. Tsolmonbaatar, T. Kaino, I. Ohtsu and H. Takagi: Vacuolar amino acid transporters upregulated by exogenous proline and involved in cellular localization of proline in Saccharomyces cerevisiae. *These authors contributed equally to this work. J. Gen. Appl. Microbiol., 62, 132-139 (2016).
- H. Takagi, J. Taguchi and T. Kaino: Proline accumulation protects Saccharomyces cerevisiae cells in the stationary phase from ethanol stress by reducing reactive oxygen species levels. Yeast, 33, 355-363 (2016).
- D. Greetham, H. Takagi and T. P. Phister: Presence of proline has a protective effect on weak acid stressed Saccharomyces cerevisiae. Anton. Leeuw. Int. J. G., 105, 641-652 (2014).
- 高木博史: 製パンプロセスにおけるパン酵母のストレス耐性:プロリン・アルギニン代謝と育種への応用. 日本食品微生物学会雑誌, 31, 185-193 (2014).
- Y. Tatehashi and H. Takagi: Characterization of γ-glutamyl kinase mutants from Saccharomyces cerevisiae. J. Biosci. Bioeng, 116, 576-579, 2013.
- Y. Sasano, Y. Haitani, K. Hashida, I. Ohtsu, J. Shima and H. Takagi: Simultaneous accumulation of proline and trehalose in industrial baker's yeast enhances fermentation ability in frozen dough. *These authors contributed equally to this work. J. Biosci. Bioeng., 113, 592-595 (2012).
- T. Kaino, Y. Tasaka, Y. Tatehashi and H. Takagi: Functional analysis of the C-terminal region of g-glutamyl kinase from Saccharomyces cerevisiae. Biosci. Biotech. Biochem., 76, 454-461 (2012).
- Y. Sasano, Y. Haitani, I. Ohtsu, J. Shima and H. Takagi: Proline accumulation in baker's yeast enhances high-sucrose stress tolerance and fermentation ability in sweet dough. Int. J. Food Microbiol., 152, 40-43 (2012).
- 高木博史: セルフクローニング法による実用パン酵母の育種:プロリン・アルギニン代謝に着目したストレス耐性の向上. 生物工学, 89, 8-12 (2011).
- T. Kaino and H. Takagi: Proline as a stress protectant in the yeast Saccharomyces cerevisiae. Biosci. Biotech. Biochem., 73, 2131-2135 (2009).
- H. Takagi: Proline as a stress protectant in yeast: physiological functions, metabolic regulations and biotechnological applications. Appl. Microbiol. Biotech., 81, 211-223 (2008).
- T. Kaino, T. Tateiwa, S. Mizukami-Murata, J. Shima and H. Takagi: Self-cloning baker's yeasts that accumulate proline enhance freeze tolerance in doughs. Appl. Environ. Microbiol., 74, 5845-5849 (2008).
- 高木博史:プロリンのストレス保護機能と代謝調節機構 酵母を用いてその魅力と謎に迫る. 化学と生物, 46, 304-306 (2008).
- T. Kaino and H. Takagi: Gene expression profiles and intracellular contents of stress protectants in Saccharomyces cerevisiae under ethanol and sorbitol stresses. Appl. Microbiol. Biotech., 79, 273-283 (2008).
- 高木博史:酵母におけるプロリンの生理機能と代謝調節機構—生物のストレス適応戦略の応用をめざして, 蛋白質核酸酵素, 53, 249-255 (2008).
- T. Sekine, A. Kawaguchi, Y. Hamano and H. Takagi: Desensitization of feedback inhibition of the Saccharomyces cerevisiae g-glutamyl kinase enhances proline accumulation and freezing tolerance. Appl. Environ. Microbiol., 73, 4011-4019 (2007).
- H. Takagi, F. Matsui, A. Kawaguchi, H. Wu, H. Shimoi and Y. Kubo: Construction and analysis of self-cloning sake yeasts that accumulate proline. J. Biosci. Bioeng., 103, 377-380 (2007).
- H. Takagi, M. Takaoka, A. Kawaguchi and Y. Kubo: Effect of L-proline on sake brewing and ethanol stress in Saccharomyces cerevisiae. Appl. Environ. Microbiol., 71, 8656-8662 (2005).
- K. Matsuura and H. Takagi: Vacuolar functions are involved in stress-protective effect of intracellular proline on Saccharomyces cerevisiae. J. Biosci. Bioeng., 100, 538-544 (2005).
- 高木博史:酵母の新しいストレス耐性機構 「異常タンパク質」の生成回避と分解. バイオサイエンスとインダストリー, 62, 672-676 (2004).
- Y. Terao, S. Nakamori and H. Takagi: Gene dosage effect of L-proline biosynthetic enzymes on L-proline accumulation and freeze tolerance in Saccharomyces cerevisiae. Appl. Environ. Microbiol., 69, 6527-6532 (2003).
- Y. Morita, S. Nakamori and H. Takagi: L-Proline accumulation and freeze tolerance in Saccharomyces cerevisiae are caused by a mutation in the PRO1 gene encoding g-glutamyl kinase. Appl. Environ. Microbiol., 69, 212-219 (2003).
- Y. Morita, S. Nakamori and H. Takagi: Effect of proline and arginine metabolism on freezing stress of Saccharomyces cerevisiae. J. Biosci. Bioeng., 94, 390-394 (2002).
- H. Takagi, K. Sakai, K. Morida and S. Nakamori: Proline accumulation by mutation or disruption of the proline oxidase gene improves resistance to freezing and desiccation stresses in Saccharomyces cerevisiae. FEMS Microbiol. Lett., 184, 103-108 (2000).
- H. Takagi, F. Iwamoto and S. Nakamori: Isolation of freeze-tolerant laboratory strain of Saccharomyces cerevisiae from proline-analogue-resistant mutants. Appl. Microbiol. Biotechnol., 47, 405-411 (1997).
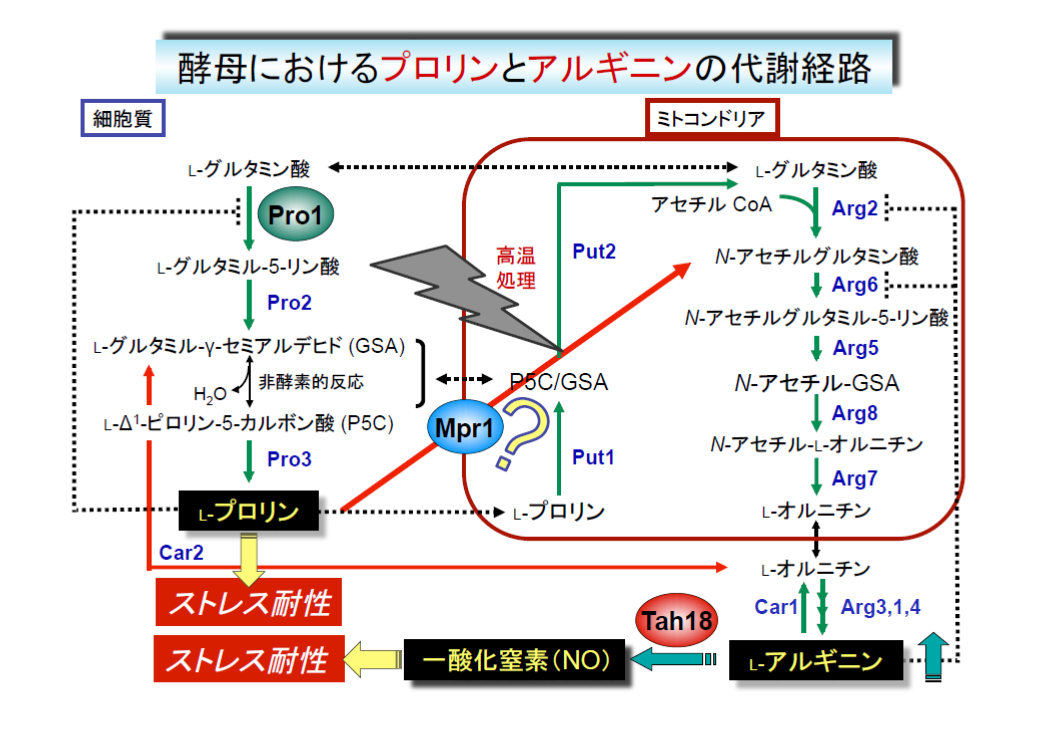
2) N-アセチルトランスフェラーゼMpr1の分子機能(プロリンからのアルギニン合成)
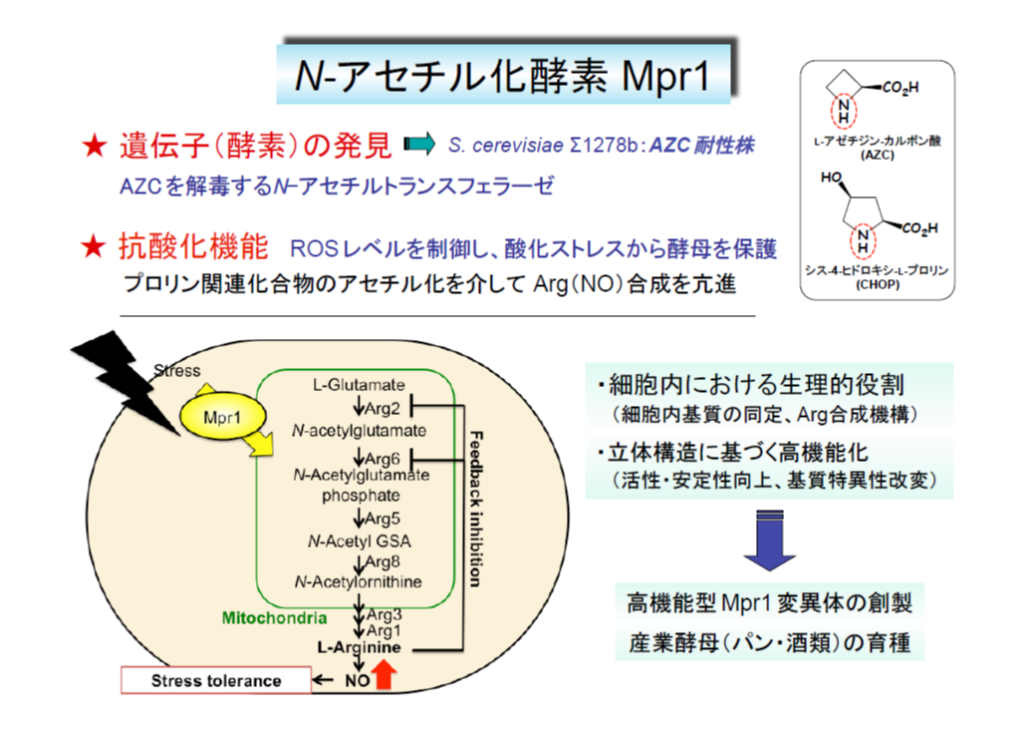
私たちはProの研究を進める過程で、S. cerevisiae Σ1278b株がAZC耐性を示すことを見出し、AZC耐性に関わる遺伝子MPR(sigma1278b gene for L-proline-analogue resistance)を単離しました。MPRはAZCをN-アセチル化し、新生タンパク質に取込まれないように解毒する酵素(Mpr1)をコードしています。MPRはS. cerevisiaeのゲノム解析株(S288C)にはありませんが、多くの酵母(分裂酵母、S. cerevisiae同胞種、パン酵母など)に存在しているため、何らかの生理機能を予想して解析を続けました。その結果、Mpr1はROSレベルが上昇する冷凍、乾燥、エタノールなどのストレスから酵母を保護することが明らかになりました。興味深いことに、パン酵母のMpr1は高温乾燥などの酸化ストレス下でROSレベルの上昇を防ぎ、発酵力に寄与しています。また、Mpr1が酸化ストレス下でアルギニン(Arg)合成を亢進することで一酸化窒素(NO)の生成を誘導し、酸化ストレス耐性に寄与している可能性を提唱しています。さらに、X線結晶構造解析によりMpr1のユニークな立体構造と反応機構を解明するとともに、ランダム変異導入や分子設計に基づき、活性・熱安定性が向上したMpr1変異体を取得、創製することができました。また、熱安定性が向上したMpr1変異体の発現により、清酒酵母の発酵力向上にも成功しました
Mpr1はこれまでの遺伝学的・酵素学的な解析から、Proまたはその代謝中間体をアセチル化し、未知の機構によりArg合成に関与することが示唆されています。現在、Mpr1を介した新規なArg合成経路と生理的意義の解明に取り組んでいます。
<発表論文>
- M. Ohashi, R. Nasuno, D. Watanabe and H, Takagi: Stable N-acetyltransferase Mpr1 improves ethanol productivity in the sake yeast Saccharomyces cerevisiae. J. Ind. Microbiol. Biotechnol.,46, 1039-1045 (2019).
- R. Nasuno, S. Hirase, S. Norifune, D. Watanabe, and H. Takagi: Structure-based molecular design for thermostabilization of N-acetyltransferase Mpr1 involved in a novel pathway of L-arginine synthesis in yeast. J. Biochem., 159, 271-277 (2016). 「JB論文賞」受賞
- 高木博史, 那須野 亮: 酵母に見出した新規な抗酸化酵素「N-アセチルトランスフェラーゼMpr1」. 化学と生物, 53, 148-155 (2015).
- R. Nasuno, Y. Hirano, T. Itoh, T. Hakoshima, T. Hibi and H. Takagi: Structural and functional analysis of the yeast N-acetyltransferase Mpr1 involved in oxidative stress tolerance via proline metabolism. Proc. Natl. Acad. Sci. USA, 110, 11821-11826 (2013).
- T. M. H. Bach and H. Takagi: Properties, metabolisms, and applications of l-proline analogues. Appl. Microbiol. Biotech., 97, 6623-6634 (2013).
- T. M. H. Bach, R. Hara, K. Kino, I. Ohtsu, N. Yoshida and H. Takagi: Microbial production of N-acetyl cis-4-hydroxy-l-proline by coexpression of the Rhizobium l-proline cis-4-hydroxylase and the yeast N-acetyltransferase Mpr1. Appl. Microbiol. Biotech., 97, 247-257 (2013).
- A. Nishimura, R. Nasuno and H. Takagi: The proline metabolism intermediate Δ1-pyrroline-5-carboxylate directly inhibits the mitochondrial respiration in budding yeast. FEBS Lett., 586, 2411-2416 (2012).
- T. M. H. Bach, T. Hibi, R. Nasuno, G. Matsuo, Y. Sasano and H. Takagi: Production of N-acetyl cis-4-hydroxy-L-proline by the yeast N-acetyltransferase Mpr1. J. Biosci. Bioeng., 114, 160-165 (2012).
- A. Nishimura, T. Kotani, Y. Sasano and H. Takagi: An antioxidative mechanism mediated by the yeast N-acetyltransferase Mpr1: Oxidative stress-induced arginine synthesis and its physiological role. FEMS Yeast Res., 10, 687-698 (2010).
- Y. Sasano*, S. Takahashi*, J. Shima and H. Takagi: Antioxidant N-acetyltransferase Mpr1/2 of industrial baker's yeast enhances fermentation ability after air-drying stress in bread dough. *These authors contributed equally to this work. Int. J. Food Microbiol., 138, 181-185 (2010).
- K. Ogawa-Mitsuhashi, K. Sagane, J. Kuromitsu, H. Takagi and K. Tsukahara: MPR1 as a novel selection marker in Saccharomyces cerevisiae. Yeast, 26, 587-593 (2009).
- 高木博史: 酵母の新規アセチルトランスフェラーゼMpr1によるプロリン代謝を介した活性酸素種の制御. 生化学, 81, 891-895 (2009).
- T. Hibi, H. Yamamoto, G. Nakamura, and H. Takagi: Crystallization and preliminary crystallographic analysis on N-acetyltransferase Mpr1 from Saccharomyces cerevisiae. Acta Crystallogr., F65, 169-172 (2009).
- K. Iinoya, T. Kotani, Y. Sasano and H. Takagi: Engineering of the yeast antioxidant enzyme Mpr1 for enhanced activity and stability. Biotechnol. Bioeng., 103, 341-352 (2009).
- T. Kotani and H. Takagi: Identification of amino acid residues essential for the yeast N-acetyltransferase Mpr1 activity by site-directed mutagenesis. FEMS Yeast Res., 8, 607-614 (2008).
- 高木博史:エタノールから酵母を守る新しいアセチル化酵素Mpr1. バイオサイエンスとインダストリー, 66, 75-77 (2008).
- M. Wada, K. Okabe, M. Kataoka, S. Shimizu, A. Yokota and H. Takagi: Distribution of L-azetidine-2-carboxylate N-acetyltransferase in yeast. Biosci. Biotech. Biochem., 72, 582-586 (2008).
- X. Du and H. Takagi: N-Acetyltransferase Mpr1 confers ethanol tolerance on Saccharomyces cerevisiae by reducing reactive oxygen species. Appl. Microbiol. Biotech., 75, 1343-1351 (2007).
- 高木博史:酵母に発見した「アセチル化酵素Mpr1」による新しい抗酸化メカニズム 活性酸素種の生成に関わる物質を基質とした酸化ストレス防御システム?!. 化学と生物, 44, 83-84 (2006).
- X. Du and H. Takagi: N-Acetyltransferase Mpr1 conferred freeze tolerance in Saccharomyces cerevisiae by reducing reactive oxygen species. J. Biochem., 138, 391-397 (2005).
- M. Nomura and H. Takagi: Role of the yeast acetyltransferase Mpr1 in oxidative stress: Regulation of oxygen reactive species caused by a toxic proline catabolism intermediate. Proc. Natl. Acad. Sci. U.S.A., 101, 12616-12621 (2004).
- T. Iwaki, N. Tanaka, H. Takagi, Y. Giga-Hama and K. Takegawa: Characterization of end4+, a gene required for endocytosis in Schizosaccharomyces pombe. Yeast, 21, 867-881 (2004).
- X.-H. Zhang, H. Takagi and J. M. Widholm: Expression of a novel yeast gene that detoxifies the proline analog azetidine-2-carboxylate confers resistance during tobacco seed germination, callus and shoot formation. Plant Cell Rep., 22, 615-622 (2004).
- M. Nomura, S. Nakamori and H. Takagi: Characterization of novel acetyltransferases found in the budding and fission yeasts that detoxifies a proline analogue, azetidine-2-carboxylic acid. J. Biochem., 133, 67-74 (2003).
- Y. Kimura, S. Nakamori and H. Takagi: Polymorphism of the MPR1 gene required for toxic proline analogue resistance in the Saccharomyces cerevisiae complex species. Yeast, 19, 1437-1445 (2002).
- M. Shichiri, C. Hoshikawa, S. Nakamori and H. Takagi: A novel acetyltranferase found in Saccharomyces cerevisiae S1278b that detoxifies a proline analogue, azetidine-2-carboxylic acid. J. Biol. Chem., 276, 41998-42002 (2001).
- 高木博史、七里美雅、中森 茂:出芽酵母Σ1278b株にプロリンアナログ耐性遺伝子を見いだした, バイオサイエンスとインダストリー, 59, 25-28 (2001).
- H. Takagi, M. Shichiri, M. Takemura, M. Mohri and S. Nakamori: Saccharomyces cerevisiae S1278b has novel gene of the N-acetyltransferase gene superfamily required for L-proline analogue resistance. J. Bacteriol., 182, 4249-4256 (2000).
3) 一酸化窒素(NO)の合成制御機構と生理的役割
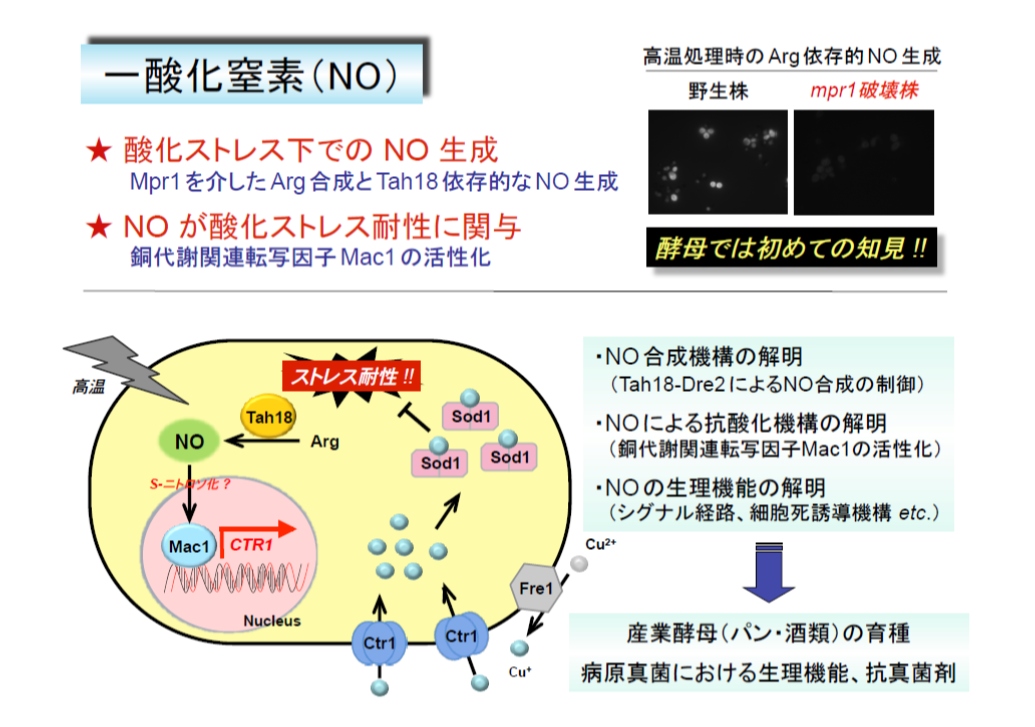
私たちは最近、S. cerevisiae Σ1278b株では、高温処理のような酸化ストレスに応答し、Mpr1を介して合成されたArgからTah18タンパク質依存的に一酸化窒素(NO)が合成され、細胞の酸化ストレス耐性に寄与することを見出しました。NOはシグナル分子として様々な生命現象に関与し、哺乳類ではArgからNO合成酵素(NOS)により生成します。一方、酵母ではゲノム上に哺乳類NOSのオルソログが存在せず、NOの研究は進んでいません。Tah18はNADPHの電子をDre2タンパク質に渡すことで細胞質の鉄硫黄タンパク質生合成に関わるジフラビン還元酵素ですが、NO合成への関与は初めての知見です。また、Dre2がTah18依存的なNOS様活性を阻害すること、酸化ストレス(過酸化水素、高温など)に応答し、Tah18-Dre2複合体が解離することから、Dre2による新規なNO合成制御機構を提唱しています。現在、酵母細胞内のNO測定系を確立するとともに、NOS活性に必要なNADPHの生成酵素およびArgの酸化に関わるオキシゲナーゼ様タンパク質の同定を進めており、NO合成制御機構の全貌を明らかにしたいと考えています。
酵母におけるNOの生理的役割についても解析しており、高温ストレス下で合成されるNOが銅代謝に関する転写因子Mac1を活性化し、銅の取込み系の亢進を介して銅依存型スーパーオキシドジスムターゼSod1活性を上昇させることで、高温に伴い生成するROSを除去し、ストレス耐性に寄与することを明らかにしました。現在、Mac1の活性化機構について、S-ニトロソ化やリン酸化などに着目して解析しています。また、NOによってS-ニトロソ化されるタンパク質やNOに応答する遺伝子を網羅的に探索し、NOの新たなシグナル伝達系の同定を試みています。一方で、過剰量のNOは細胞死を誘導することから、NOの機能二面性(細胞保護・細胞死)を提唱しています。また、パン酵母において高機能型のPro1やMpr1を発現させると、NOレベルの増加とともに、ストレス後のROSレベルの低下、細胞生存率や発酵力の向上が見られました。今後、効率良くNOを合成することで、ストレス耐性が向上した産業酵母の育種に繋げたいと考えています。さらに、分裂酵母Schizosaccharomyces pombeにおいても、NOの合成系や解毒酵素の同定、酸化ストレス下におけるNOを介したシグナル伝達系の存在を実証しました。
一方、真菌感染症の原因となる病原真菌(Candida glabrata, Cryptococcus neoformans, Aspergillus fumigatus)においても、ゲノム上にMpr1やTah18などのオルソログ遺伝子が存在しています。これらの病原真菌はヒトに感染する際、温度・低酸素などのストレスに応答して耐性を獲得し、病原性を示すことから、病原真菌におけるNOの分子機能について解析を進めています(千葉大学真菌医学研究センター、筑波大学との共同研究)
2019年度には、NOの機能二面性の発見が学術的に高く評価され、酵母や糸状菌におけるNOの分子機能の解明とその応用をめざす研究が科研費基盤研究(S)に新規採択されました。
https://www.jsps.go.jp/j-grantsinaid/12_kiban/ichiran_r01/j-data/r_1_jp_19h05639.pdf
https://www.jsps.go.jp/j-grantsinaid/12_kiban/ichiran_r01/shoken/r_1_shinsa_19h05639.pdf
さらに、NO研究から発展し、2019年10月に着任した西村 明助教が所属していた赤池孝章教授(東北大医学系研究科)のグループは、真核生物が活性イオウ分子であるシステインパースルフィドを利用することで、酸素やNADHに依存しないエネルギー代謝「イオウ呼吸」を営んでいることを発見しました。そこで、当研究室ではイオウ呼吸の詳細なメカニズムを解明するため、真核生物のモデルである酵母を用いて解析を進めています。また、得られた知見を活用し、酵母による有用物質の生産性向上やミトコンドリア病の治療薬開発を試みる予定です(赤池教授との共同研究)。
https://www.jstage.jst.go.jp/article/kagakutoseibutsu/56/8/56_560803/_pdf/-char/ja
<発表論文>
- Y. Yoshikawa, R. Nasuno, N. Takaya and H. Takagi: Metallothionein Cup1 attenuates nitrosative stress in the yeast Saccharomyces cerevisiae. Microbial Cell, 10, 170-177 (2023).
- S. Shino, R. Nasuno and H. Takagi: S-Glutathionylation of fructose-1,6-bisphosphate aldolase confers nitrosative stress tolerance on yeast cells via a metabolic switch. Free Radic. Biol. Med., 193, 319-329 (2022).
- S. Oiki, R. Nasuno, S. Urayama, H. Takagi and D. Hagiwara: Intracellular production of reactive oxygen species and a DAF-FM-related compound in Aspergillus fumigatus in response to antifungal agent exposure. Sci. Rep., 12, 13516 (2022).
- R. Nasuno, Y. Yoshikawa and H. Takagi: Acetaldehyde reacts with a fluorescence nitric oxide probe harboring o-phenylenediamine structure interfering with fluorometry. Free Radic. Biol. Med., 187, 29-37 (2022).
- S. Eknikom, R. Nasuno and H. Takagi: Molecular mechanism of ethanol fermentation inhibition via protein tyrosine nitration of pyruvate decarboxylase by reactive nitrogen species in yeast. Sci. Rep., 12, 4664 (2022).
- R. Nasuno, S. Suzuki, S. Oiki, D. Hagiwara and H. Takagi: Identification and functional analysis of GTP cyclohydrolase II in Candida glabrata in response to nitrosative stress. Front. Microbiol., 13, 825121 (2022)
- R. Nasuno, N. Yoshioka, Y. Yoshikawa and H. Takagi: Cysteine residues in the fourth zinc finger are important for activation of the nitric oxide-inducible ranscription factor Fzf1 in the yeast Saccharomyces cerevisiae. Genes Cells, 26, 823-829 (2021).
- R. Nasuno, N. Iwai and H. Takagi: Development of a microtiter plate-based analysis method of nitric oxide dioxygenase activity. J. Gen. Appl. Microbiol., J. Gen. Appl. Microbiol., 68, 38-41 (2022).
- Y. Yoshikawa, R. Nasuno and H. Takagi: NADPH is important for isobutanol tolerance in a minimal medium of Saccharomyces cerevisiae. Biosci. Biotech. Biochem., 85, 2084-2088 (2021).
- Y. Yoshikawa, R. Nasuno and H. Takagi: An NADPH-independent mechanism enhances oxidative and nitrosative stress tolerance in yeast cells lacking glucose-6-phosphate dehydrogenase activity. Yeast, 38, 414–423 (2021).
- R. Nasuno, Y. Yoshikawa and H. Takagi: The analytical method to identify the nitrogen source for nitric oxide synthesis. Biosci. Biotech. Biochem., 85, 211-214 (2021). DOI: 10.1093/bbb/zbaa046.
- R. Nasuno, S. Shino, Y. Yoshikawa, N. Yoshioka, Y. Sato, K. Kamiya and H. Takagi: Detection system of the intracellular nitric oxide in yeast by HPLC with a fluorescence detector. Anal. Biochem., 598, 113707 (2020). DOI: 10.1016/j.ab.2020.113707
- K. Anam, R. Nasuno and H. Takagi: A novel mechanism for nitrosative stress tolerance dependent on GTP cyclohydrolase II activity involved in riboflavin synthesis of yeast. Sci. Rep., 10, 6015, DOI: 10.1038/s41598-020-62890-3 (2020).
- A. Nishimura, R. Nasuno, Y. Yoshikawa, M. Jung, T. Ida, T. Matsunaga, M. Morita, H. Takagi, H. Motohashi and T. Akaike: Mitochondrial cysteinyl-tRNA synthetase is expressed via alternative transcriptional initiation regulated by energy metabolism in yeast. J. Biol. Chem., J. Biol. Chem., 294, 13781-13788 (2019).
- 那須野 亮, 吉川雄樹, 高木博史: 酵母における一酸化窒素シグナルを介したストレス応答機構. 生化学, 90, 701-705 (2018).
- D. Hagiwara*, H. Takahashi, H. Takagi, A. Watanabe and K. Kamei: Heterogeneity in pathogenicity-related properties and stress tolerance in Aspergillus fumigatus clinical isolates.Med. Mycol. J., 59, E63-E70 (2018).
- R. I. Astuti*, R. Nasuno* and H. Takagi: Nitric oxide signaling in yeast. *These authors contributed equally to this work. Adv. Microb. Physiol., 72, 29-63 (2018).
- 那須野 亮, 吉川雄樹, 高木博史: 酵母に見出した一酸化窒素(NO)の合成制御機構と生理機能. 化学と生物, 55, 617-623 (2017).
- 高木博史: 酵母における一酸化窒素の分子機能と応用. バイオサイエンスとインダストリー, 75, 214-218 (2017).
- R. I. Astuti*, R. Nasuno*, and H. Takagi: Nitric oxide signaling in yeast. *These authors contributed equally to this work. Appl. Microbiol. Biotech., 100, 9483-9497 (2016).
- Y. Yoshikawa*, R. Nasuno*, N. Kawahara, A. Nishimura, D. Watanabe, and H. Takagi: Regulatory mechanism of the flavoprotein Tah18-dependent nitric oxide synthesis and cell death in yeast. *These authors contributed equally to this work. Nitric Oxide-Biol. Chem., 57, 85-91 (2016).
- R. I. Astuti*, D. Watanabe*, and H. Takagi: Nitric oxide signaling and its role in oxidative stress response in Schizosaccharomyces pombe. *These authors contributed equally to this work. Nitric Oxide-Biol. Chem., 52, 29-40 (2016).
- R. Nasuno, M. Aitoku, Y. Manago, A. Nishimura, Y. Sasano and H. Takagi: Nitric oxide-mediated antioxidative mechanism in yeast through the activation of the transcription factor Mac1. PLoS One, 9(11), e113788 (2014).
- D. Watanabe, R. Kikushima, M. Aitoku, A. Nishimura, I. Ohtsu, R. Nasuno and H. Takagi: Exogenous addition of histidine reduces copper availability in the yeast Saccharomyces cerevisiae. Microbial Cell, 1, 241-246 (2014).
- H. Kitagaki and H. Takagi: Mitochondrial metabolism and stress response of yeast: Applications in fermentation technologies. J. Biosci. Bioeng., 117, 383-393 (2014).
- 高木博史: 一酸化窒素を介した酵母の新しい抗酸化メカニズムとその応用. バイオサイエンスとインダストリー, 71, 343-345 (2013).
- A. Nishimura, N. Kawahara and H. Takagi: The flavoprotein Tah18-dependent NO synthesis confers high-temperature stress tolerance on yeast cells. Biochem. Biophys. Res. Commun., 430, 137-143 (2013).
- Y. Sasano*, Y. Haitani*, K. Hashida, I. Ohtsu, J. Shima and H. Takagi: Enhancement of the proline and nitric oxide synthetic pathway improves fermentation ability under multiple baking-associated stress conditions in industrial baker's yeast. *These authors contributed equally to this work. Microb. Cell Fact., 11:40 doi:10.1186/1475-2859-11-40 (2012).
2. ユビキチンシステムによる異常タンパク質の修復・分解機構
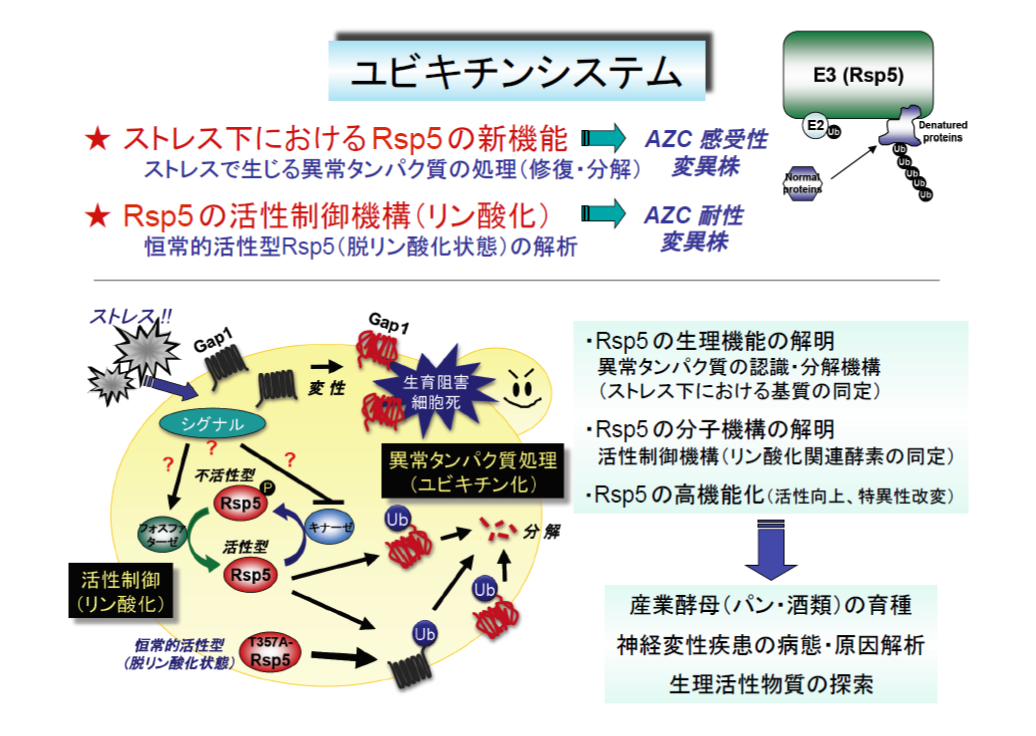
ユビキチン(Ub)システムは、真核生物の細胞内で役目が終わり不用になったタンパク質やストレスで生じる異常タンパク質の処理だけでなく、様々な細胞機能の制御に重要な役割を担っています。Ubシステムにおいて、Ubリガーゼは標的タンパク質に結合し、分解の目印であるUb分子を連結する重要な酵素です。
本研究は、野生型株が生育する低濃度のAZCを加えた培地でも生育できない「AZC超感受性変異株」の分離が出発です。変異株ではRsp5の遺伝子に変異(Ala401Glu)が入っており、Rsp5の基質であるGap1パーミアーゼが分解されず、細胞膜上で安定に活性を維持するため、過剰のAZCが細胞内に取込まれ、超感受性になることが判明しました。また、この変異株は多くのストレス(栄養培地での高温培養、酸化、エタノール、浸透圧、イオンなど)に対しても感受性を示すことから、Rsp5が「ストレスで生じる異常タンパク質の処理」に関与するというモデルを提唱しています。これまでの研究から、Rsp5がストレス転写因子Msn2/4の発現制御(RNA分子の核外輸送など)を介してストレスタンパク質の発現を調節し、異常タンパク質の修復に関与すると考えています。さらに、Egd2やPda1などのタンパク質がストレス下でRsp5依存的にユビキチン化を受け、分解されることから、Rsp5は異常タンパク質の分解にも関わっていると考えられます。
私たちは、知見の乏しい細胞膜タンパク質の品質管理についても、アミノ酸パーミアーゼGap1をモデル基質として解析しています。その結果、エタノール存在下でGap1のアミノ酸取込み活性は顕著に低下し、Rsp5によってUb化されることが判明しました。興味深いことに、NH4+の添加に伴うUb化とは異なるパターンのUb化も観察されました。このようにGap1は環境変化に応答し、異なるアダプタータンパク質に認識され、Ub化されることから、ストレス下における特異的なタンパク質品質管理機構が存在することを示しました。また、Rsp5の基質特異性を生み出す新たな機構として、3つのWWドメイン内の保存されたThr残基のリン酸化による制御を見出し、Rsp5による基質認識の特異性・多様性の一端を明らかにしています。
さらに、ストレス耐性の向上をめざす応用研究にも取り組んでいます。実験室酵母ではRsp5とUb結合酵素(Ubc1-13)を同時に過剰発現させると、様々なストレス耐性が向上しました。また、WWドメインへのランダム変異導入で得られたRsp5変異体(Thr255Ala, Thr357Ala)は酢酸ナトリウムやAZCに対する耐性が向上しました。産業酵母では、Rsp5の過剰発現により高濃度醸造に資するビール酵母を作製し、バイオエタノール酵母でThr255Ala変異体を発現させると、酢酸ナトリウム存在下での発酵力が向上しました。最近では、パン酵母が冷凍後も高い発酵力を維持する仕組みとして、「Ub-プロテアソーム系」が冷凍により変性したタンパク質の分解に寄与することを見出しました(テーブルマーク(株)との共同研究)。
https://bsw3.naist.jp/research/index.php?id=1645
http://www.naist.jp/news/2018/04/004990.html
その他、基質タンパク質の認識に関与するWWドメイン内にランダム変異を導入し、パーキンソン病など神経変性疾患の原因タンパク質として知られるヒトα-シヌクレインの過剰発現による毒性を軽減するRsp5の変異体を複数取得しました(JB論文賞)。特に、Pro343Ser変異体ではα-シヌクレインのUb化を介した分解が促進されていました。一方、Thr255Ala変異体はα-シヌクレインの分解には影響ありませんが、細胞の酢酸耐性を向上させ、酢酸存在下でのエタノール発酵が改善されました。以上の結果から、Rsp5のWWドメインが異常タンパク質認識の特異性・多様性を生み出すことで、酵母は様々なストレスに対する適応能力を獲得すると考えられます。
また、Rsp5自身の活性制御機構にも興味を持っており、細胞にAZC耐性を付与する恒常活性型変異体(Thr357Ala)の解析から、Rsp5では基質認識ドメインのリン酸化による立体障害が基質認識能を変化させ、活性を制御するという仮説を立て検証しています。
このように、ストレスにおけるUbシステムの機能を解明するとともに、Ubシステムを強化し、ストレス耐性を向上させる手法を「Ubシステム工学」と名付け、産業酵母への応用を目指しています。さらに、発酵化学産業だけでなく、創薬・治療への応用を目指した共同研究も進めています(岩手大学・Chulalongkorn University(タイ):新規な生理活性物質の探索、Danube Private University(オーストリア):神経変性疾患の病態・原因の解析)。
<発表論文>
- A. Sangkaew, T. Kojornna, R. Tanahashi, H. Takagi and C. Yompakdee: A novel yeast-based screening system for potential compounds that can alleviate human α-synuclein toxicity. J. Appl. Microbiol., 132, 1409-1421 (2022).
- R. Tanahashi, T. Matsushita, A. Nishimura and H. Takagi: Downregulation of the broad-specificity amino acid permease Agp1 mediated by the ubiquitin ligase Rsp5 and the arrestin-like protein Bul1 in yeast. Biosci. Biotech. Biochem., 85, 1266-1274 (2021).
- R. Tanahashi, T. S. N.Afiah, A. Nishimura, D. Watanabe and H. Takagi: The C2 domain of the ubiquitin ligase Rsp5 is required for ubiquitination of the endocytic protein Rvs167 upon change of nitrogen source. FEMS Yeast Res., 20, foaa058(2020). DOI: 10.1093/femsyr/foaa058.
- J. Yoshida, Y. Okawa, T. Oyama, S. Uesugi, H. Takagi, Y. Ito, and K. Kimura: Inhibition of calcineurin and glycogen synthase kinase-3β by ricinoleic acid derived from castor oil. Lipids, 55, 89-99 (2020).
- 渡辺大輔, 高木博史: 酵母ユビキチンリガーゼRsp5による選択的な基質認識とその応用への可能性 ユビキチンの「誤送信」を防ぐ. 化学と生物, 57, 36-42 (2018).
- A. Watcharawipas, D. Watanabe and H. Takagi*: Sodium acetate responses in Saccharomyces cerevisiae and the ubiquitin ligase Rsp5. Front. Microbiol., 9, 2495 (2018).
- D. Watanabe, H. Sekiguchi, Y. Sugimoto, A. Nagasawa, N. Kida and H. Takagi: Importance of proteasome gene expression during model dough fermentation after freezing preservation of baker’s yeast cells. Appl. Environ. Microbiol., 84(12), e00406-18 (2018).
- C. T. Mai, Q. G. Le, Y. Ishiwata-Kimata, H. Takagi, K. Kohno and Y. Kimata: 4-Phenylbutyrate suppresses the unfolded protein response without restoring protein folding in Saccharomyces cerevisiae. FEMS Yeast Res., FEMS Yeast Res., 18, foy016 (2018).
- A. Watcharawipas, D. Watanabe and H. Takagi: Enhanced sodium acetate tolerance in Saccharomyces cerevisiae by the Thr255Ala mutation of the ubiquitin ligase Rsp5. FEMS Yeast Res., DOI: 10.1093/femsyr/fox083 (2017).
- D. Watanabe, H. Murai, R. Tanahashi, K. Nakamura, T. Sasaki and H. Takagi: Cooperative and selective roles of the WW domains of the yeast Nedd4-like ubiquitin ligase Rsp5 in the recognition of the arrestin-like adaptors Bul1 and Bul2. Biochem. Biophys. Res. Commun., 463, 76-81 (2015).
- I. Wijayanti*, D. Watanabe*, S. Oshiro and H. Takagi: Isolation and functional analysis of yeast ubiquitin ligase Rsp5 variants that alleviate the toxicity of human a-synuclein. *These authors contributed equally to this work. J. Biochem., 157, 251-260 (2015). 「JB論文賞」受賞
- S. Oshiro and H. Takagi: The transcriptional activator Pog1 controls cell cycle and its phosphorylated form is downregulated by the ubiquitin ligase Dma2 in Saccharomyces cerevisiae. FEMS Yeast Res., 14, 1015-1027 (2014).
- T. Shiga, N. Yoshida, Y. Shimizu, E. Suzuki, T. Sasaki, D. Watanabe and H. Takagi: Quality control of plasma membrane proteins by yeast Nedd4-like ubiquitin ligase Rsp5p under environmental stress conditions. Eukaryot. Cell, 13, 1191-1199 (2014).
- S. Uesugi, D. Watanabe, M. Kitajima, R. Watanabe, Y. Kawamura, M. Ohnishi, H. Takagi and K. Kimura: Calcineurin inhibitors suppress the high-temperature stress sensitivity of the yeast ubiquitin ligase Rsp5 mutant: A new method of screening for calcineurin inhibitors. FEMS Yeast Res., 14, 567-574 (2014).
- T. Sasaki and H. Takagi: Phosphorylation of a conserved Thr357 in yeast Nedd4-like ubiquitin ligase Rsp5 is involved in downregulation of the general amino acid permease Gap1. Genes Cells, 18, 459-475 (2013).
- T. Ogata, H. Yamagishi, K. Ukibe and H. Takagi:Construction of bottom-fermenting yeasts that overexpress the ubiquitin ligase RSP5 gene and their effects on high-gravity fermentation. J. Brew. Distill., 3, 1-5 (2012).
- 高木博史: ストレスにおける酵母のユビキチンリガーゼRsp5の役割とその応用. 化学と生物, 49, 100-107 (2011).
- H. Hiraishi, T. Shimada, I. Ohtsu, T. Sato and H. Takagi: The yeast ubiquitin ligase Rsp5 down-regulates the alpha subunit of nascent polypeptide-associated complex Egd2 under stress conditions. FEBS J., 276, 5287-5297 (2009).
- H. Hiraishi, M. Okada, I. Ohtsu and H. Takagi: Functional analysis of the yeast ubiquitin ligase Rsp5: Involvement of the ubiquitin-conjugating enzyme Ubc4 and poly-ubiquitination in ethanol-Induced down-regulation of targeted proteins. Biosci. Biotech. Biochem., 73, 2268-2273 (2009).
- Y. Haitani, M. Nakata, T. Sasaki, A. Uchida and H. Takagi: Engineering of the yeast ubiquitin ligase Rsp5: isolation of a new variant that induces constitutive inactivation of the general amino acid permease Gap1. FEMS Yeast Res., 9, 73-86 (2009).
- Y. Haitani and H. Takagi: Rsp5 is required for the nuclear export of mRNA of HSF1 and MSN2/4 under stress conditions in Saccharomyces cerevisiae. Genes to Cells, 13, 105-116 (2008).
- M. Demae, Y. Murata, M. Hisano, Y. Haitani, J. Shima and H. Takagi: Overexpression of two transcriptional factors, Kin28 and Pog1, suppresses the stress sensitivity caused by the rsp5 mutation in Saccharomyces cerevisiae. FEMS Microbiol. Lett., 277, 70-78 (2007).
- H. Hiraishi, M. Mochizuki and H. Takagi: Enhancement of stress tolerance in Saccharomyces cerevisiae by overexpression of ubiquitin ligase Rsp5 and ubiquitin-conjugating enzymes. Biosci. Biotech. Biochem., 70, 2762-2765 (2006).
- Y. Haitani, H. Shimoi and H. Takagi: Rsp5 regulates expression of stress proteins via post-translational modification of Hsf1 and Msn4 in Saccharomyces cerevisiae. FEBS Lett., 580, 3433-3438 (2006).
- H. Takagi: Analysis and application of degradation mechanism of abnormal proteins by the yeast Rsp5 ubiquitin ligase. Report of the Noda Institute for Scientific Research, 49, 78-80 (2005).
- 高木博史:酵母ユビキチンリガーゼの新しい機能 ストレスで生じる異常タンパク質分解への関わり. 化学と生物, 42, 432-434 (2004).
- C. Hoshikawa, M. Shichiri, S. Nakamori and H. Takagi: A non-conserved Ala401 in the yeast Rsp5 ubiquitin ligase is involved in degradation of Gap1 permease and stress-induced abnormal proteins. Proc. Natl. Acad. Sci. U.S.A., 100, 11505-11510 (2003).
3.ストレス耐性機構の高機能化と高度利用による産業酵母の育種
私たちは高等生物のモデルとして重要な酵母を用いて、独自に見出した「ストレス耐性機構」を分子レベルで解明し、得られた成果を発酵食品・バイオエタノールの製造に使用されている産業酵母の育種への応用にも積極的に取り組んでいます(例えば、農研機構・生研センターの大型競争的資金を連続して獲得し、共同研究を推進してきました)。
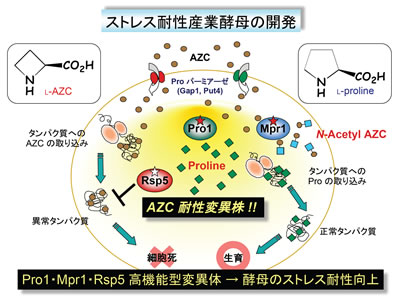
上記の1や2で紹介した「Pro蓄積に関与するPro1」、「酸化ストレスに伴うNO合成に関与するMpr1」、「異常タンパク質処理に関与するRsp5」の各遺伝子に適切な変異が導入された酵母は、AZC耐性が向上し、かつストレス耐性の向上も期待できます。したがって、遺伝子組換え技術ではなく、古典的手法(突然変異処理)を用いてAZC耐性変異株を分離することで、同様の機構によってストレス耐性の向上した産業酵母の育種も可能でしょう。今後、パン酵母と泡盛酵母について、実用化をめざした研究を進めていきます。
((独)農業・食品産業技術総合研究機構 生物系特定産業技術研究支援センター「新技術・新分野創出のための基礎研究推進事業」採択課題(H16-20度)
「酵母の発酵環境ストレス適応機構の解明と新規な発酵生産系開発への基盤研究」
(http://brain.naro.affrc.go.jp/tokyo/marumoto/up/h16kadai/03takagi.htm)
(http://brain.naro.affrc.go.jp/tokyo/marumoto/up/h20seika/03takagi.htm)
発酵生産環境において酵母には多様なストレスが負荷され、細胞内タンパク質の変性に伴う異常タンパク質の生成により有用機能が制限されます。本研究では、研究代表者等が見出した「異常タンパク質の生成回避・検知処理機構」を中心に酵母のストレス適応機構を解明します。また、実用ストレス条件下における酵母の遺伝子発現ネットワークを解明し、ストレス適応機構の理解に役立てます。さらに、各機構の高機能化と高度利用により、優れたストレス耐性を有する産業酵母(パン酵母、清酒酵母)の作製と実用化に向けた開発技術を確立します。
本研究で得られた酵母のストレス適応機構や実用ストレス環境における産業酵母の遺伝子情報に関する知見を活用し、高度なストレス耐性産業酵母の開発が期待できます。例えば、プロリンやMpr1は冷凍生地やドライイーストの効率的生産を可能にします。また、製パンストレスと酸化ストレスとの共通性もパン酵母の育種に応用できます。さらに、エタノール高生産と耐性に関する成果は、有用な清酒酵母やバイオエタノール酵母の開発に寄与できます((独)食総研、(独)酒総研との共同研究)。
((独)農業・食品産業技術総合研究機構 生物系特定産業技術研究支援センター「イノベーション創出基礎的研究推進事業 発展型研究(一般枠)」採択課題(H21-23度)
「酸化ストレス耐性酵母の創製と革新的発酵生産システムの開発」
(http://brain.naro.affrc.go.jp/tokyo/marumoto/inv_up/press090702/24takagi.htm)
酵母の発酵生産環境は細胞内に活性酸素種(ROS)を蓄積する酸化ストレス状態であり、既存の酵母は酸化ストレスに脆弱であることが、発酵能を制限し、酵母の効率的な利用を妨げている最大要因です。私たちは新規な酸化ストレス耐性機構を見出し、耐性向上のための機構解明や技術構築をほぼ完了しています。しかし、酵母の耐性向上技術を実用に供するためには、高度な酸化ストレス耐性を付与した産業酵母の創製と革新的な発酵生産システムの開発を行なう必要があります。具体的には、酸化ストレス耐性機構(適合溶質の蓄積、抗酸化タンパク質の発現、タンパク質分解系の強化)の高機能化と高度利用を行ない、産業酵母の耐性向上のための育種戦略を構築します。また、有用育種技術のセルフクローニング法により、高度な酸化ストレス耐性を示す産業酵母(パン酵母、酒類酵母、バイオエタノール酵母)を作製し、実用に即した条件で特性評価を行ない、発酵生産システムを構築します。
本研究で行う酸化ストレス耐性酵母の創製により、製パン分野では、長期保存冷凍生地、超高糖生地、高耐久性乾燥酵母などの製造技術を構築できます。酒類・バイオエタノール製造分野では、高濃度エタノールの生産や発酵時間の短縮が期待できます((独)食総研、アサヒビール(株)、(独)酒総研との共同研究)。
<発表論文>
- H. T. Phuong, Y. Ishiwata-Kimata, Y. Nishi, N. Oguchi, H. Takagi and Y. Kimata: Aeration mitigates endoplasmic reticulum stress even without mitochondrial respiration in Saccharomyces cerevisiae. Microbial Cell, 8, 77-86 (2021).
- H. Takagi: Molecular mechanisms and highly-functional development for stress tolerance of the yeast Saccharomyces cerevisiae. Biosci. Biotech. Biochem., 85, 1017-1037 (2021).
- Y. Nabilah Binti Mohd Fauzee, N. Taniguchi, Y. Ishiwata-Kimata, H. Takagi and Y. Kimata*: The unfolded protein response in Pichia pastoris without external stressing stimuli. FEMS Yeast Res., 20, foaa053(2020). DOI: 10.1093/femsyr/foaa053.
- M. E. Prastya, R. I. Astuti, I. Batubara, H. Takagi and A. T. Wahyudi: Natural extract and its fractions isolated from the marine bacterium Pseudoalteromonas flavipulchra STILL-33 have antioxidant and antiaging activities in Schizosaccharomyces pombe. FEMS Yeast Res., 20, foaa014 (2020). DOI: 10.1093/femsyr/foaa014.
- M. E. Prastya, R. I. Astuti, I. Batubara, H. Takagi and A. T. Wahyudi: Chemical screening identifies an extract from marine Pseudomonas sp.-PTR-08 as an anti-aging agent that promotes fission yeast longevity by modulating the Pap1-ctt1+ pathway and the cell cycle. Mol. Biol. Rep., 47, 33-43 (2020).
- D. A. Nur’utami, L. Haditjaroko, H. Takagi, K. Syamsu: Hyperosmotic stress tolerance of transcription activator Msn2-Over expression strain and proline-NO synthesis strain of Saccharomyces cerevisiae in very high gravity bioethanol fermentation. Pak. J. Biotechnol., 14, 135-139 (2017).
- S. Hirayama, M. Shimizu, N. Tsuchiya, S. Furukawa, D. Watanabe, H. Shimoi, H. Takagi, H. Ogihara, and Y. Morinaga: Awa1p on the cell surface of sake yeast inhibits biofilm formation and the co-aggregation between sake yeasts and Lactobacillus plantarum ML11-11. J. Biosci. Bioeng., 119, 532-537 (2015).
- H. Takagi, K. Hashida, D. Watanabe, R. Nasuno, M. Ohashi, T. Iha, M. Nezuo, and M. Tsukahara: Isolation and characterization of awamori yeast mutants with l-leucine accumulation that overproduce isoamyl alcohol. J. Biosci. Bioeng., 119, 140-147 (2015).
- T. Inaba, D. Watanabe, Y. Yoshiyama, K. Tanaka, J. Ogawa, H. Takagi, H. Shimoi and J. Shima: An organic acid-tolerant HAA1-overexpression mutant of an industrial bioethanol strain of Saccharomyces cerevisiae and its application to the production of bioethanol from sugarcane molasses. AMB Express, 3:74, doi:10.1186/2191-0855-3-74 (2013).
- H. Kitagaki and H. Takagi: Mitochondrial metabolism and stress response of yeast: Applications in fermentation technologies. J. Biosci. Bioeng., 117, 383-393 (2014).
- Y. Sasano, Y. Haitani, K. Hashida, S. Oshiro, J. Shima and H. Takagi: Improvement of fermentation ability under baking-associated stress conditions by altering the POG1 gene expression in baker's yeast. Int. J. Food Microbiol., 165, 241-245 (2013).
- T. Inai, D. Watanabe, Y. Zhou, R. Fukada, T. Akao, J. Shima, H. Takagi and H. Shimoi: Rim15p-mediated regulation of sucrose utilization during molasses fermentation using Saccharomyces cerevisiae strain PE-2. J. Biosci. Bioeng, 116, 591-594, 2013.
- S. Hasegawa, T. Ogata, K. Tanaka, A. Ando, H. Takagi and J. Shima: Overexpression of vacuolar H+-ATPase-related genes in bottom-fermenting yeast enhances ethanol tolerance and fermentation rates during high-gravity fermentation. J. Inst. Brew., 118, 179-185 (2012).
- 高木博史: 酵母の発酵環境ストレス耐性機構の解析と実用酵母の育種への応用. 「発酵・醸造食品の最新技術と機能性II」(監修 北本勝ひこ), pp.50-59 (2011).
- Y. Sasano*, Y. Haitani*, K. Hashida, I. Ohtsu, J. Shima and H. Takagi: Overexpression of the transcription activator Msn2 enhances fermentation ability of industrial baker’s yeast in frozen dough. *These authors contributed equally to this work. Biosci. Biotech. Biochem., 76, 624-627 (2012).
- Y. Sasano*, D. Watanabe*, K. Ukibe, T. Inai, I. Ohtsu, H. Shimoi and H. Takagi: Overexpression of the yeast transcription activator Msn2 confers furfural resistance and increases the initial fermentation rate in ethanol production. *These authors contributed equally to this work. J. Biosci. Bioeng., 113, 451-455 (2012).
- H. Urbanczyk, C. Noguchi, H. Wu, D. Watanabe, T. Akao, H. Takagi, H. Shimoi: Sake yeast strains have difficulty in entering a quiescent state after cell growth cessation. J. Biosci. Bioeng., 112, 44-48 (2011).
- T. Nakamura, S. Takahashi, H. Takagi and J. Shima: Multicopy suppression of oxidant-sensitive eos1 mutation by IZH2 in Saccharomyces cerevisiae and the involvement of Eos1 in zinc homeostasis. FEMS Yeast Res., 10, 259-269 (2010).
- K. Ukibe, K. Hashida, N. Yoshida and H. Takagi: Metabolic engineering of Saccharomyces cerevisiae for astaxanthin production and oxidative stress tolerance. Appl. Environ. Microbiol., 75, 7205-7211 (2009).
- S. Takahashi*, A. Ando*, H. Takagi and J. Shima: Insufficiency of copper ion homeostasis causes freeze-thaw injury of yeast cells revealed by indirect gene expression analysis. *These authors contributed equally to this work. Appl. Environ. Microbiol., 75, 6706-6711 (2009).
- J. Shima and H. Takagi: Stress-tolerance of baker's yeast (Saccharomyces cerevisiae) cells: stress-protective molecules and genes involved in stress tolerance. Biotechnol. Appl. Biochem., 53, 155-164, 2009.
- H. Wu, T. Watanabe, Y. Araki, H. Kitagaki, T. Akao, H. Takagi and H. Shimoi: Disruption of ubiquitin-related genes in laboratory yeast strains enhances ethanol production during sake brewing. J. Biosci. Bioeng., 107, 636-640, 2009.
- T. Nakamura, H. Takagi and J. Shima: Effects of ice-seeding temperature and intracellular trehalose contents on survival of frozen Saccharomyces cerevisiae cells. Cryobiol., 58, 170-174 (2009).
- Y. Araki, H. Wu, H. Kitagaki, T. Akao, H. Takagi and H. Shimoi: Ethanol stress stimulates the Ca2+-mediated calcineurin/Crz1 pathway in Saccharomyces cerevisiae. J. Biosci. Bioeng., 107, 1-6 (2009).
- T. Nakamura, S. Mizukami-Murata, A. Ando, Y. Murata, H. Takagi and J. Shima: Changes in gene expression of commercial baker's yeast during an air-drying process that simulates dried yeast production. J. Biosci. Bioeng., 106, 405-408 (2008).
- J. Shima, A. Ando and H. Takagi: Possible roles of vacuolar H+-ATPase and mitochondrial function in tolerance to air-drying stress revealed by genome-wide screening of Saccharomyces cerevisiae deletion strains. Yeast, 25, 179-190 (2008).
- F. Tanaka-Tsuno, S. Mizukami-Murata, Y. Murata, T. Nakamura, A. Ando, H. Takagi and J. Shima: Functional genomics of commercial baker's yeasts that have different abilities for sugar utilization and high-sucrose tolerance under sugar conditions. Yeast, 24, 901-911 (2007).
- T. Nakamura, A. Ando, H. Takagi and J. Shima: Eos1, whose deletion confers sensitivity to oxidative stress, is involved in N-glycosylation in Saccharomyces cerevisiae. Biochem. Biophys. Res. Commun. 353, 293-298 (2007).
- A. Ando, T. Nakamura, Y. Murata, H. Takagi and J. Shima: Identification and classification of genes required for tolerance to freeze-thaw stress revealed by genome-wide screening of Saccharomyces cerevisiae deletion strains. FEMS Yeast Res., 7, 244-253 (2007).
- H. Wu, X. Zheng, Y. Araki, H. Sahara, H. Takagi and H. Shimoi: Global gene expression analysis of yeast cells during sake brewing. Appl. Environ. Microbiol., 72, 7353-7358 (2006).
- F. Tanaka, A. Ando, T. Nakamura, H. Takagi and J. Shima: Functional genomic analysis of commercial baker's yeast during initial stages of model dough-fermentation. Food Microbiology, 23, 717-728 (2006).
- M. Sugiura and H. Takagi: Yeast cell death caused by mutation of the OST2 gene encoding the e-subunit of the Saccharomyces cerevisiae oligosaccharyltransferase. Biosci. Biotech. Biochem., 70, 1234-1241 (2006).
- A. Ando, F. Tanaka, Y. Murata, H. Takagi and J. Shima: Identification and classification of genes required for tolerance to high sucrose stress revealed by genome-wide screening of Saccharomyces cerevisiae. FEMS Yeast Research, 6, 249-267 (2006).
- M. Wada, S. Nakamori and H. Takagi: Serine racemase homologue of Saccharomyces cerevisiae has L-threo-3-hydroxyaspartate dehydratase activity. FEMS Microbiol. Lett., 225, 189-193 (2003).
- Y. Kubo, H. Takagi and S. Nakamori: Effect of gene disruption of succinate dehydrogenase on succinate production in a sake yeast strain. J. Biosci. Bioeng., 90, 619-624 (2000).

