成果報告
論文No.008
コンテキストデータおよびマルチプラットフォームメタボロミクスデータ解析のための代謝物識別名の統合化
- Redestig Henning、草野都、福島敦史、松田史生、斎藤和季、有田正規
- BMC Bioinformatics 11, 214 (2010)
Redestig, H., Kusano, M., Fukushima, A., Matsuda, F., Saito, K. and Arita, M. (2010) Consolidating metabolite identifiers to enable contextual and multi-platform metabolomics data analysis. BMC Bioinformatics 11: 214
高速分析から得られるデータ解析は、検出された生体化合物によって記述されるデータがいかに整理されているかに依存する。遺伝子や転写物、タンパク質のメタデータはよく整理されているが、代謝物については未整理のままである。化合物識別名はその用途によって様々なものがあり、一貫性がない。オンラインの化合物データベースは様々なタイプの識別名を併記して使用しているが、これらの統合化はなされていない。そこで我々はローカルおよび公共データベースから代謝物識別名を統合化する手法とそれを実装したソフトウェアを開発した。統合に際し、本法はいわゆる1つのプライマリ識別名にだけ依存するのではない。また本プログラムは代謝物データを集めた SQLiteデータベースから得られる化合物および代謝物の識別名を内部統合した構造からなる。作成されたデータベースを用いて、自前の代謝物識別名とKEGGのような外部のデータベースから得られる類義語のつながりを調べることができる。また、それらを調査するためのクエリは、コマンドラインおよび統計プログラミング言語Rで実行可能である。代謝物識別名の相互参照は、メタボロミクスデータ解析の鍵である。これを実行するために、我々はオープンソースソフトウェアmetabolite masking tool (MetMask)を開発した。本ソフトウェアはこれまで述べたアイデアを実装している。
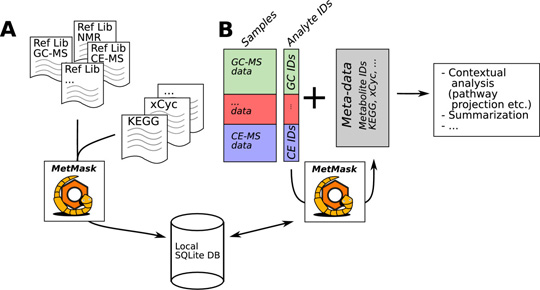
図1 MetMaskの概念図
MetMaskによる代謝物識別名の統合化。(A) ローカルデータベースは検出化合物や代謝物をリスト化した参照ライブラリ(Re lib)および公共データベースをインポートすることにより構築される。(B)構築したデータベースにより識別名およびメタデータが迅速に抽出できる。これらはデータの集約化や生合成経路への投影といったコンテキスト分析に使用可能である。
|
