研究成果の紹介
DNA polymerase IV(Pol IV)の新たな機能の解明この論文はJ Biol Chem.誌のPapers of the Weekに選ばれました
大腸菌の染色体DNA複製は主としてDNA Polymerase III (Pol III)が行っている。しかし鋳型DNA鎖上にUVや薬剤などによる損傷を受けた塩基があると、このポリメラーゼはDNA合成を停止してしまう。そこで、損傷乗り越えDNA合成(translesion synthesis, TLS)ポリメラーゼと呼ばれる特殊なDNAポリメラーゼが損傷部位を合成し、DNA複製反応を継続させると考えられている。しかし、このDNAポリメラーゼの交代(ポリメラーゼスイッチ反応)がどのようなメカニズムで起こるかはよく解っていない。この研究では、大腸菌のTLSポリメラーゼの一つであるPol IVを用いて、試験管内でPol IIIからPol IVへのポリメラーゼスイッチ反応を再構成し、その過程を詳細に調べた。この研究からPol IVの新たな活性が見いだされ、ポリメラーゼスイッチ反応がどのように起こるかについての重要な可能性が示された。
Pol IIIはβ sliding clamp(β clamp、図中リング状タンパク質)と結合してDNA 上をスライドしながら連続的なDNA合成を行なっており、DNA合成を停止してもすぐには鋳型DNAからは外れないと考えられている。停止したPol III からTLSポリメラーゼへの交代が起こる際、TLSポリメラーゼがどのようにPol IIIによって保持された鋳型DNAの3'-プライマー末端にアクセスするかは大きな問題である。この研究では、精製したポリメラーゼを使った試験管内DNA複製系を利用し、実際に停止したPol IIIからPol IVへのポリメラーゼスイッチ反応が起こるかどうかを調べた。その結果、極めて短時間のうちにスイッチ反応が起き、新たにPol IIIと交代したPol IVが3'-プライマー末端からDNA合成反応を開始した。また、このポリメラーゼスイッチ反応と同時にPol III がβ clampから外れることを見いだし、Pol IVには能動的にPol IIIをβ clampから外す活性があることを示した。この系では、二種類のポリメラーゼが極めて短時間の間に次々と3'-プライマー末端から外れては結合する事を繰り返していると考えられる。この現象は今回見つかったPol IVの新しい活性によるものであり、Pol IVはPol IIIとβ clampとの結合を阻害する事によりポリメラーゼスイッチ反応を促進しているのではないかと考えている。
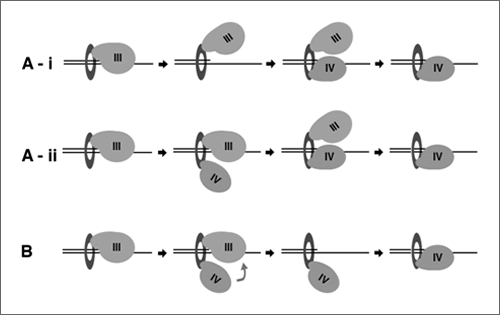
図 ポリメラーゼスイッチ反応のモデル Aではポリメラーゼスイッチ反応に続いてPol IIIのβ clampからの解離が起こるモデルを2パターン示す。BではPol IVはPol IIIをβ clampから外すことにより3'-プライマー末端をアクセス可能な状態にし、ポリメラーゼスイッチ反応を起こしている。
掲載論文
Furukohri A, Goodman MF, Maki H. (2008) A Dynamic Polymerase Exchange with Escherichia coli DNA Polymerase IV Replacing DNA Polymerase III on the Sliding Clamp. J Biol Chem. 283(17), 11260-9.
(2008年04月01日掲載)
